Алис Е. Полард
1 MRC Лондонски институт по медицински науки, Imperial College London, болница Hammersmith, Лондон W12 0NN, Великобритания
2 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Cambridge, UK
Луис Мартинс
1 MRC Лондонски институт по медицински науки, Imperial College London, болница Хамерсмит, Лондон W12 0NN, Великобритания
Филип Дж. Мъкет
1 MRC Лондонски институт по медицински науки, Imperial College London, болница Hammersmith, Лондон W12 0NN, Великобритания
Санджай Хадайат
1 MRC Лондонски институт по медицински науки, Imperial College London, болница Hammersmith, Лондон W12 0NN, Великобритания
Орели Борно
2 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Cambridge, UK
Мариам Клаузен
3 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Гьотеборг, Швеция
Тереза Адмира
3 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Гьотеборг, Швеция
Микаел Бюрсел
3 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Гьотеборг, Швеция
Ребека Фиадейро
1 MRC Лондонски институт по медицински науки, Imperial College London, болница Hammersmith, Лондон W12 0NN, Великобритания
Лора Уилсън
1 MRC Лондонски институт по медицински науки, Imperial College London, болница Hammersmith, Лондон W12 0NN, Великобритания
Чад Уайлдинг
1 MRC Лондонски институт по медицински науки, Imperial College London, болница Hammersmith, Лондон W12 0NN, Великобритания
Василиос Н. Котиадис
4 Катедра по клетъчна и биология на развитието и UCL консорциум за митохондриални изследвания, Университетски колеж Лондон, Лондон, Великобритания
Майкъл Р. Дюшен
4 Катедра по клетъчна и биология на развитието и UCL консорциум за митохондриални изследвания, Университетски колеж Лондон, Лондон, Великобритания
Даниел Сътън
5 Лекарствена безопасност и метаболизъм, IMED Biotech Unit, AstraZeneca, Babraham, CB22 3AT
Люси Пенфолд
1 MRC Лондонски институт по медицински науки, Imperial College London, болница Hammersmith, Лондон W12 0NN, Великобритания
Алесандро Сардини
1 MRC Лондонски институт по медицински науки, Imperial College London, болница Hammersmith, Лондон W12 0NN, Великобритания
Мохамад Бохлули-Й
3 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Гьотеборг, Швеция
Дейвид М. Смит
2 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Cambridge, UK
Джон А. Прочетете
2 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Cambridge, UK
Майкъл А. Сноудън
2 Discovery Sciences, IMED Biotech Unit, AstraZeneca, Cambridge, UK
Анджела Уудс
1 MRC Лондонски институт по медицински науки, Imperial College London, болница Хамерсмит, Лондон W12 0NN, Великобритания
Дейвид Карлинг
1 MRC Лондонски институт по медицински науки, Imperial College London, болница Hammersmith, Лондон W12 0NN, Великобритания
6 Институт за клинични науки, Имперски колеж в Лондон, болница Хамърсмит, Лондон W12 0NN, Великобритания
Свързани данни
Наличност на данни
Наборите от данни, които подкрепят констатациите от това проучване, са достъпни от съответния автор при поискване. Наборите от данни за РНК-последователност, използвани в това проучване, са достъпни от Gene Expression Omnibus (GEO) с номер за присъединяване> GSE120429.
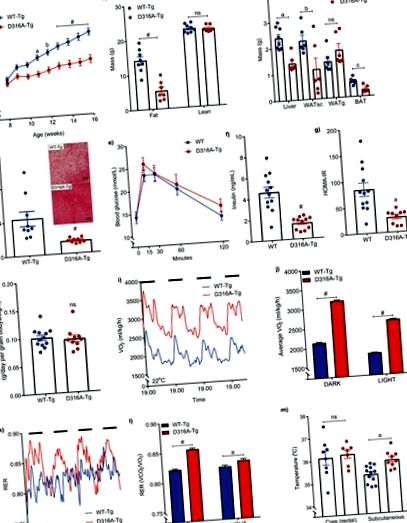
Мъжки мишки, експресиращи или див тип γ1 (WT-Tg), или D316A γ1 трансген (D316A-Tg) са били хранени с диета с високо съдържание на мазнини (HFD) от 8-седмична възраст. а, Тегло (n = 20 за WT-Tg и 17 за D316A-Tg). a P = 0,002, b P = 0,0006 и # P # P a P = 0,0004, b P = 0,0378, c P = 0,0012, ns = без значение). д, Нива на чернодробни триглицериди при мишки, хранени с HFD в продължение на 16 седмици (n = 9 на генотип; # P # P a P = 0.0012). з, Прием на храна за 5-дневен период (n = 12 за WT-Tg и 10 за D316A-Tg, ns = незначителен). i, Консумацията на кислород в цялото тяло (VO2) се наблюдава непрекъснато за период от 84 часа и (j) средно VO2 през 12 часа светли и 12 часа тъмни периоди (тъмни периоди, представени от плътните черни ленти). Точките от данни от отделни мишки бяха пропуснати в графиката, за да покажат по-ясно средните стойности и размерите на лентата за грешка. # P a P = 0,0086 # P a P = 0,0022, ns = без значение). Във всички случаи показаните резултати са средната стойност ± sem. Статистическите анализи в панели a, b, e, j и l бяха извършени чрез двупосочен ANOVA, последван от тест за множество сравнения на Bonferroni. Статистическите анализи в панели c, f, g, h и m бяха извършени чрез t-тест на Student, несдвоени, двустранни, с корелация на Welch, приложена към f и g. Данните в панел d бяха анализирани чрез тест на Ман-Уитни.
Анализът на RNAseq е използван за определяне на промени в генната експресия в WATsc, изолирани от мишки WT-Tg и D316A-Tg, хранени с HFD в продължение на 16 седмици (n = 6 мишки на генотип). а, Вулканичен сюжет с регулирани надолу гени, показани в синьо, регулирани нагоре в червено и непроменени в сиво. 1353 гена бяха регулирани нагоре и 1102 гена бяха регулирани надолу (промяна в пъти> 1,5). Идентифицирани са диференциално експресирани гени (DEG) (P a P = 0,0026 и b P = 0,0494; ns = не е значимо. е, Вулканичен сюжет като в (а) от WATsc от мишки, хранени с чау диета (n = 6 мишки за генотип). 1361 гена бяха регулирани нагоре и 1808 гена бяха регулирани надолу. Atp2a1 и Ryr1 са подчертани в сюжета. ж, Показани са най-значително обогатените анотации на генна онтология (GO) за значително регулирани гени. з, Схематична диаграма, показваща регулирани гени (подчертани в червено) при гликолиза и TCA цикъл в D316A WATsc при мишки, хранени с чау-диета. Резултатите, показани в панел e, са средната стойност ± sem и статистически анализ, извършен чрез множество t-тестове, коригирани за множество сравнения.
За разлика от промените в генната експресия, наблюдавани при HFD, транскриптомният анализ на WATsc от мишки, хранени с диета, разкрива поразително различен модел на експресия. Над 3000 гена бяха значително променени (промяна в пъти> 1,5, P Фиг. 4f). Гените в пътищата, участващи в метаболизма на мастните киселини, TCA цикъла, производството на митохондриални АТР и гликолизата, бяха силно обогатени с WATsc от мишки D316A-Tg (фиг. 4g, h), докато гените, свързани със скелетната мускулатура, включително Atp2a1 и Ryr1, бяха намалени. -регулиран (фиг. 4е). Взети заедно, тези резултати показват, че при HFD, но не и при диета с чау, активирането на AMPK предизвиква превключване в WATsc към фенотип, подобен на скелетни мускули. Обратно, при диета с чау, активирането на AMPK води до промени в експресията на гени, участващи в катаболните пътища, включително гликолиза и TCA цикъл.
Нашите открития показват, че активирането на AMPK увеличава дела на кафявоподобните адипоцити в депото на WATsc, но тези клетки се различават от конвенционалните бежови адипоцити, тъй като те не експресират Ucp1. Преминаването на мишки от чау към диета с високо съдържание на мазнини води до значителна промяна в транскрипционния отговор в тези кафяви клетки, което води до повишена експресия на гени, свързани със скелетните мускули. Предлагаме, че тези адипоцити, посочени като скелетно мускулни AMPK, препрограмирани термогенни (SMART) адипоцити, могат да представляват неразпознат досега клетъчен тип или фенотип, който обикновените подкожни бели адипоцити могат да постигнат по време на трайно активиране на AMPK. Елегантни проучвания, използващи техники за проследяване на родословието, разкриват, че различни видове адипоцити могат да се развият от един и същ пул на предшественици, които пораждат скелетни мускулни клетки 16, 25, 26. Съвременните данни сочат, че миоцитите и кафявите адипоцити произлизат от Myf5 + -предшественик, докато белите адипоцити произлизат от различен предшественик. Повечето проучвания показват, че бежовите адипоцити произлизат от бял адипоцитен предшественик (Myf5 -), произтичащ главно от депото WATsc и тяхното развитие се увеличава в отговор на адаптация на студ 16, 25, 26 .
Методи
Животни
Метаболитен фенотип
Лечение с β-гуанидинопропионова киселина
В някои случаи мишките бяха прехвърлени на HFD и след 1 седмица им беше даден достъп до вода, съдържаща 0,5% β-гуанидинопропионова киселина и 0,13% захарин или вода, съдържаща само 0,13% захарин. Водата, приема на храна и телесното тегло са били наблюдавани през следващите 3 седмици.
Измерване на дишането в експланти от бяла мастна тъкан
Подкожната бяла мастна тъкан (1,5-2 mg) се получава с помощта на биопсичен перфоратор (2 mm) и се поставя в XF24 Islet Capture Microplates и се инкубира в пробна среда (DMEM без субстрат (Sigma D5030), 30 mM NaCl, pH7,4, 25 тМ глюкоза, 0,5 тМ натриев пируват). Скоростта на консумация на кислород беше измерена на анализатор Sehaorse XF24 Flux след последователно добавяне на олигомицин (5 μM), карбонил цианид 3-хлорофенил хидразон (CCCP, 5 μM) и антимицин (5 μM).
Хистологичен анализ
Western blot анализ
Тъканите се хомогенизират, като се използва Ultra-Turrax хомогенизатор в 10x w/v ледено студен буфер за хомогенизиране, съдържащ 50 mM Tris, 50 mM NaF, 5 mM Napyrophosphate, 1 mM EDTA, 0.25 M манитол, 1 mM дитиотреитол, 157 μg/ml бензамидин, 4 μg/ml трипсинов инхибитор и 0,1 тМ фенилметилсулфонил флуорид. Хомогенатите се центрофугират при 13 000 х g в продължение на 15 минути за отстраняване на неразтворим материал. Съдържанието на протеин в разтворимата фракция се определя количествено, като се използва BCA комплект за анализ (ThermoSciaching). Протеините (общо 50 μg) се разделят чрез SDS-PAGE и се прехвърлят в поливинилиден дифлуоридна мембрана (Millipore Immobilon-FL) при 100 V за 90 минути. Мембраните се оцветяват с PonceauS за проверка на протеиновия трансфер и се блокират в 4% (w/v) говежди серумен албумин (BSA) за 1 час при стайна температура. Освен ако не е посочено друго, първичните антитела се разреждат 1: 1000 в TBS, съдържащи 4% BSA и 0,1% Tween-20, и се инкубират с мембраната в продължение на 4 часа при стайна температура или за една нощ при 4 ° С. Мембраните се промиват интензивно с TBS, съдържащ 0,1% Tween-20 преди инкубация с подходящо вторично антитяло IRDye (LI-COR Biosciences) в TBS за 1 час при стайна температура. Блотите се визуализират с помощта на системата за изображения на Odyssey (LI-COR Biosciences) и се определят количествено с помощта на ImageStudio 4.0.
Антитела
В това проучване са използвани следните антитела: общ коктейл от антитела OXPHOS (Abcam, ab110413); Ckmt2 (Abcam, ab55963); Флаг (клетъчно сигнализиране, 14793); Pgc1-a (Abcam, ab54481); Том-20 (SantaCruz, sc-11415); Serca1 (Abcam, ab109899); Serca2 (Invitrogen, MA3-919); Ucp1 (Abcam, ab 10983); винкулин (Sigma, V9131).
Изолация на РНК
Обща РНК се изолира от бързо замразена тъкан чрез хомогенизиране в 1 ml TRIzol (Life Technologies) на 100 mg тъкан върху лед. Пробите се съхраняват една нощ при -20 ° С, центрофугират се при 10 000 х g в продължение на 15 минути и хомогенатът се отстранява в прясна епруветка. Прибавя се хлороформ (400 ml на ml) и сместа се центрофугира при 10 000 х g в продължение на 15 минути при стайна температура. Водната фаза се прехвърля в без РНКаза Eppendorf и се добавя абсолютен етанол (0,53 х обем). РНК се пречиства с помощта на RNeasy Mini спин колони (Qiagen). РНК се елуира в 50 μl свободна от RNase H2O и се определя количествено с помощта на NanoDrop спектрофотометър. Пробите се замразяват при -80 ° С, докато се наложи.
RT-qPCR
3 μg РНК се инкубира с 1 μl произволни хексамери (50 ng/μl), 1 μl 10 mM dNTP смес и DEPC H2O до 10 μl. Пробите се инкубират при 65 ° С в продължение на 5 минути на термоциклер. 10 μl cDNA синтезна смес (съдържаща 2 μl 10 x RT буфер, 4 μl 25 mM MgCl2, 2 μl 0,1 M DTT, 1 μl RNaseOut и 1 μl Superscript II (Qiagen)) се добавя към всяка проба и се циклира, както следва: 25 ° С (10 минути), 85 ° С (50 минути) и епруветките се прехвърлят върху лед, докато се охладят. 1 μl RnaseH се добавя към всяка проба и се инкубира при 37 ° С в продължение на 10 минути. За да се определи линейността на праймера, 5 μl кДНК от всяка проба се събират и серийно се разреждат, за да се получи стандартна крива. За всяка PCR реакция се добавят 5 μl кДНК към 10 μl 2x SYBR-HiROX (Bioline), 1,6 μl напред (AGCCATCTGCATGGGATCAAA) и обратно (GGGTCGTCCCTTTCCAAAGTG) праймер смес и ddH2O до общ реакционен обем от 20 μl. Плочата qPCR беше анализирана с помощта на Opticon термоциклер със софтуер за монитор Opticon за генериране на c (t) стойности за всяка реакция.
За количествено определяне на генната експресия, c (t) повторения на стойността бяха проверени за ефективност на праймера (%) и консистенция. След това се изчисляват средни стойности на c (t) за всяка проба и се определят количествено, като се използва линейно уравнение (y = mx + c), предварително определено от стандартната крива, съответстващо на гена от интерес. Уравнението беше решено за x, с y като стойност c (t). След това тази стойност се трансформира с помощта на 10 х, тъй като стандартната крива се генерира в логаритмична скала. Този процес се повтаря за всички гени, включително определен ген за домакинство. Освен ако не е посочено друго, всички експерименти са нормализирани до Polr2a, РНК полимераза, за която е доказано, че остава стабилна при диетични интервенции.
След това генната експресия беше представена като съотношение към експресията на домакинския ген за експеримента и като кратна промяна спрямо определената контрола.
РНК-последователен анализ
РНК се екстрахира от подкожно бяло мастно вещество (6 мишки на генотип, хранени или с чау, или с HFD в продължение на 16 седмици, обработени индивидуално). За всяка проба 2 μg РНК в 60 μl без RNase H2O беше оценка на качеството, използвайки комплект BioAnalyzer RNA (Agilent CA, САЩ). За допълнителен анализ се изисква оценка на RNA Integrity Number (RIN)> 7. РНК библиотеките бяха подготвени с помощта на TruSeq Stranded mRNA Library Prep Kit (Illumina) и стандартен протокол Illumina. Количествените библиотеки се определят с Qubit HS (ThermoFisher) и Agilent BioAnalyzer, коригирани до подходящата концентрация за секвениране. Индексираните библиотеки бяха обединени и секвенирани при крайна концентрация от 1.6 рМ на високоефективен цикъл Illumina NextSeq 500, използвайки химия на сдвоени краища с дължина на четене 75 bp. Данните за секвениране бяха демултиплексирани с помощта на Illumina bcl2fastq2-v2.16. Качеството на четенията беше оценено с помощта на FastQC (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/). Четенията бяха обработени и картографирани в генома на мишката mm10 с помощта на Bcbio-nextgen framework версия 0.9.0 (https://github.com/chapmanb/bcbio-nextgen). Използваният алайнер беше STAR 2.4.1d и качеството на подравняването беше оценено с QualiMap v.2.1.1.
Идентифицирането на диференциално експресирани гени беше извършено с помощта на DESeq2 в R. Основен компонентен анализ беше генериран като стъпка за контрол на качеството, за да се оцени групирането на проби по отношение на генната експресия. Генериран е списък с диференциално експресирани гени (DEG), изразени като Log2 кратна промяна върху контролната проба (WT-Tg), с коригирана P стойност за всеки ген.
Анализ на диференциално експресирани гени (DEG)
Статистически анализ
Освен ако не е посочено друго, представените данни са показани средно ± sem. За графики и статистически анализ е използван софтуер Graphpad Prism (v7). За сравнение между две групи, наборите от данни бяха анализирани чрез t-тестове на Student, със статистическа значимост, определена като P стойност от (74K, pdf)
Допълнителна информация
Благодарности
Тази работа е финансирана от Съвета за медицински изследвания във Великобритания (предоставяне на MC_US_A654_0003 на DC). AEP се финансира от награда за студентство BBSRC-CASE (BB/L502662/1). LW беше финансиран от награда за студентство на British Heart Foundation. Бихме искали да благодарим на членовете на екипа по физиология на цели животни (MRC LMS) за тяхното съдействие.
Бележки под линия
Наличност на данни
Наборите от данни, които подкрепят констатациите от това проучване, са достъпни от съответния автор при поискване. Наборите от данни за РНК-последователност, използвани в това проучване, са достъпни от Gene Expression Omnibus (GEO) с номер за присъединяване> GSE120429.
Резюме на отчета
Допълнителна информация за изследователския дизайн е достъпна в Резюмето за докладване на изследванията на природата, свързано с тази статия.
Принос на автора
AEP, LM, PJM, TA, MB, LW и RF извършиха in vivo и in vitro проучвания. SK, AB и MC извършиха RNAseq и анализа. CW и DS помогнаха при имунохистохимичния анализ. AS и MBY помогнаха за метаболитното фенотипизиране. LP и AW помогнаха при фенотипния анализ на мишките. AEP, LM, WK и MRD извършиха изследванията на клетъчното дишане. AEP, LM, AW, DMS, JAR, MAS и DC са проектирали и планирали проучването. Всички автори са допринесли за подготовката на ръкописа.
Конкуриращи се интереси
Авторите не декларират конкуриращи се интереси.
- Животински модел на болестта на Кашин-Бек, предизвикан от диета с ниско хранене и излагане на токсин Т-2 -
- Здраве на предците, затлъстяване и смърфове - Диетичен лекар
- Ползите от здравословното хранене са по-големи при хора с висок генетичен риск от затлъстяване Генетично
- Апоптоза, предизвикана от нисковъглехидратна и високобелтъчна диета при черен дроб на плъхове
- Модел на мишка за хроничен панкреатит, предизвикан от диета с алкохол и високо съдържание на мазнини