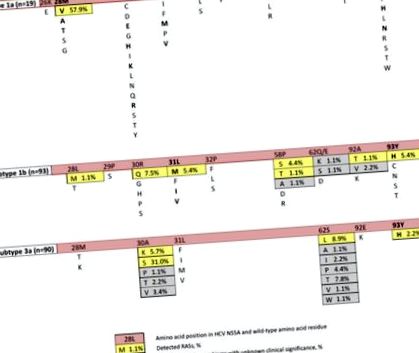
Пълен списък на аминокиселинните полиморфизми във вируса на хепатит С (HCV) NS5A, свързани с резистентност към директно действащи антивирусни средства (DAA). Свързаните с резистентност замествания (RAS) с удебелен шрифт са избрани при> 10% от вирусологичните откази (въз основа на публикуваните данни [3, 4, 5, 7, 9, 13, 18, 19].
Филогенетично дърво за HCV 1a NS5A последователности (1126 нуклеотида, позиции 6330–7455 според HCV 1a референтен щам H77 (GenBank NC004102)), изградено по GTR модел; клонове с групова надеждност> 90% са обозначени в червено; Последователностите на HCV 1а от това проучване са обозначени в синьо; типът RAS (M28V) или див тип (WT) са посочени за всяка последователност. HCV1a последователностите от Япония [20] са обозначени в зелено. Последователности, изолирани от интравенозните потребители на наркотици, маркирани като IDU.
Аминокиселинни ковариационни мрежи за NS5A последователности, изградени за HCV 1b без RAS (n = 72) (a); HCV 1b с поне един RAS (n = 21) (b); HCV 3a без RAS (n = 51) (c); HCV 3a с поне един RAS (n = 39) (d); HCV 1a (n = 19) (e); HCV 1a от Япония (n = 26) (f). Ковариациите на аминокиселините в рамките на подравняването на HCV последователностите се изобразяват с ковариращите позиции (възли), представени като кръгове, а ковариациите между позициите (ръбовете) като линии. Ковариансните мрежи бяха изградени с помощта на алгоритъм, наречен „Fastcov“, внедрен в езика за програмиране golang (https://golang.org) (https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4958985/). Изпълнимите двоични файлове са достъпни на http://yanlilab.github.io/fastcov. Мрежите бяха визуализирани с помощта на пакета R igraph.
Епитопични профили на див тип NS5A на HCV 1a, 1b и 3a, предсказващи локализация на CD8 + Т клетъчни епитопи в руската (а) и „глобална“ (в) последователности, и CD4 + Т клетъчните епитопи на руски (д) и „ глобални “(g) последователности; Разлика в епитопичните резултати на HCV 1a, 1b и 3a със и без RAS в RAS-приютиращия регион между aa 20 до 100 за CD8 + Т клетъчни епитопи в руски (b) и „глобални“ (d) последователности и CD4 + T клетъчни епитопи в руски (f) и „глобални“ (h) последователности, позициите на RAS са подчертани на криви в диаманти. Прогнозите бяха направени с помощта на NetMHCpan-4.0 и NetMHCIIpan-3.2 и се основаваха на набор от широко разпространени в световен мащаб HLA-A, HLA-B, HLA-C и HLA-DR молекули, извлечени от алелната честотна база данни и 5 HLA-DP и 6 HLA-DQ молекули. Профилите на епитопа са конструирани за всеки подтип на HCV чрез изчисляване на броя на предвидените HLA свързващи пептиди, припокриващи тази позиция, претеглени от честотата на дадената HLA и нормализирани така, че максималната стойност да е 1 при всеки цикъл на анализ.
Резюме
Пълен списък на аминокиселинните полиморфизми при вируса на хепатит С (HCV) NS5A, свързани с резистентност към директно действащи антивирусни средства (DAA). Свързаните с резистентност замествания (RAS) с удебелен шрифт са избрани при> 10% от вирусологичните откази (въз основа на публикуваните данни [3, 4, 5, 7, 9, 13, 18, 19].
Филогенетично дърво за HCV 1a NS5A последователности (1126 нуклеотида, позиции 6330–7455 според HCV 1a референтен щам H77 (GenBank NC004102)), изградено по GTR модел; клонове с групова надеждност> 90% са обозначени в червено; Последователностите на HCV 1а от това проучване са обозначени в синьо; типът RAS (M28V) или див тип (WT) са посочени за всяка последователност. HCV1a последователностите от Япония [20] са обозначени в зелено. Последователности, изолирани от интравенозните потребители на наркотици, маркирани като IDU.
Аминокиселинни ковариационни мрежи за NS5A последователности, изградени за HCV 1b без RAS (n = 72) (a); HCV 1b с поне един RAS (n = 21) (b); HCV 3a без RAS (n = 51) (c); HCV 3a с поне един RAS (n = 39) (d); HCV 1a (n = 19) (e); HCV 1a от Япония (n = 26) (f). Ковариациите на аминокиселините в рамките на подравняването на HCV последователностите се изобразяват с ковариращите позиции (възли), представени като кръгове, а ковариациите между позициите (ръбовете) като линии. Ковариансните мрежи са изградени с помощта на алгоритъм, наречен „Fastcov“, внедрен в езика за програмиране golang (https://golang.org) (https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4958985/). Изпълнимите двоични файлове са достъпни на http://yanlilab.github.io/fastcov. Мрежите бяха визуализирани с помощта на пакета R igraph.
Епитопични профили на див тип NS5A на HCV 1a, 1b и 3a, предсказващи локализация на CD8 + Т клетъчни епитопи в руската (а) и „глобална“ (в) последователности, и CD4 + Т клетъчните епитопи на руски (д) и „ глобални “(g) последователности; Разлика в епитопичните резултати на HCV 1a, 1b и 3a със и без RAS в RAS-приютиращия регион между aa 20 до 100 за CD8 + Т клетъчни епитопи в руски (b) и „глобални“ (d) последователности и CD4 + T клетъчни епитопи в руски (f) и „глобални“ (h) последователности позициите на RAS са подчертани на криви в диаманти. Прогнозите бяха направени с помощта на NetMHCpan-4.0 и NetMHCIIpan-3.2 и се основаваха на набор от широко разпространени в световен мащаб HLA-A, HLA-B, HLA-C и HLA-DR молекули, извлечени от алелната честотна база данни и 5 HLA-DP и 6 HLA-DQ молекули. Профилите на епитопа са конструирани за всеки подтип на HCV чрез изчисляване на броя на предвидените HLA свързващи пептиди, припокриващи тази позиция, претеглени от честотата на дадената HLA и нормализирани така, че максималната стойност да е 1 при всеки цикъл на анализ.
- Стандартизация на безплатни пълнотекстови методи за биомедицини за провеждане на проучвания за вродени цветове
- Пълнотекстов майчински произход и разнообразие от хаплотип на седем руски кози популации
- Биомолекули Безплатен пълнотекстов диетичен нитрат от сок от цвекло за хипертония Систематичен преглед
- Безплатен пълнотекстов намален местен отговор на кортикостероиди при еозинофилни хронични биомолекули
- Безплатни пълноценни хранителни предпочитания за животни при котки Ефект от диетичния състав и присъщи