Свързани данни
Резюме
В настоящата работа са получени рибозоми, събрани в бактериални клетки в отсъствието на основен рибозомен протеин L5. След спиране на синтеза на L5, клетките на Escherichia coli се разделят ограничен брой пъти. През това време се случва натрупване на дефектни големи рибозомни субединици. Тези 45S частици нямат повечето от компонентите на централната издатина (CP) (5S rRNA и протеини L5, L16, L18, L25, L27, L31, L33 и L35) и не са в състояние да се свържат с малката рибозомна субединица. В същото време 5S рРНК се намира в цитоплазмата в комплекс с рибозомни протеини L18 и L25 в количества, равни на количеството рибозоми. По този начин е първата демонстрация, че протеинът L5 играе ключова роля за формирането на CP по време на сглобяването на голямата рибозомна субединица в бактериалната клетка. С оглед на получените данни се обсъжда възможен модел за CP сглобяване in vivo.
ВЪВЕДЕНИЕ
Реконструкцията на функционално активни бактериални рибозомни субединици е извършена за първи път успешно преди повече от 40 години (1–3) и оттогава е натрупано голямо количество експериментални данни относно рибозомното сглобяване in vitro (4–7). Вече са налични кристални структури на рибозомите, както и тези на функционалните рибозомни комплекси от няколко бактерии, които дават представа за пространствената организация на сглобената рибозома (8–13).
В тази работа ние изучихме ролята на L5 в рибозомния сбор in vivo. Получихме щам Е. coli, където хромозомният ген, кодиращ r-протеин L5 (rplE), беше инактивиран в присъствието на комплементиращ плазмид, който осигурява индуцируема експресия на rplE. По този начин синтезът на L5 може да бъде спрян чрез отстраняване на индуктора от растежна среда. Анализът на рибозомите от L5-изчерпани клетки показва натрупване на големи рибозомни субединици, на които липсват повечето от CP компонентите. Нашите данни показват за първи път, че r-протеинът L5 играе ключова роля за формирането на целия този структурен домейн на бактериалната рибозома in vivo.
МАТЕРИАЛИ И МЕТОДИ
Щамове, плазмиди и бактериологични техники
Всички щамове и плазмиди, споменати в настоящата работа, са изброени в допълнителна таблица S1. Клетките се култивират при 37 ° С в LB бульон и LB-агарови плаки, допълнени (където е посочено) със 100 μg/ml ампицилин, 20 μg/ml хлорамфеникол и 0,2% (тегл./Об.) 1 -арабиноза. За да се получи rplE-кодиращ комплементарен плазмид pNK12, NdeI-EcoRI фрагментът от pKAB122, носещ E. coli rplE отворена рамка за четене, беше клониран в плазмида pBADET. Последното е подарък от А. Калиман. Това е производно на pBAD18 (33), вж. Допълнителна таблица S1 за повече информация. По този начин pNK12 носи rplE под контрола на индуцируем от арабиноза промотор и придава устойчивост на ампицилин. Делецията на хромозомния rplE ген се извършва чрез прехвърляне на алел ΔrplE: cat от KNB219 към щама от див тип W3110, трансформиран с pNK12, чрез генерализирана трансдукция от фаг P1 съгласно (34). Трансдуктантите са избрани върху LB агар, допълнен с хлорамфеникол и арабиноза. За простота наричаме получения щам MS01, което означава, че той винаги съдържа плазмида pNK12.
Условия за култивиране на клетки
За да се получат клетки на Е. coli, изчерпани за протеин L5, щам MS01 се отглежда при 37 ° C в LB среда, допълнена с ампицилин и арабиноза до A600 ∼ 0,4. След това клетките се измиват и инокулират с 15–20 vol предварително затоплена при 37 ° C LB среда и се култивират, докато кривата на растеж достигне плато (при A600 ∼ 0,8–1, в рамките на 6–8 часа). През това време са взети аликвотни части от клетъчната култура за седиментационен анализ на рибозомите. Клетките бяха събрани, измити с буфер (30 mM Tris – HCl, рН 7.5, 200 mM NH4Cl, 10 mM MgCl2, 0.1 mM етилендиаминтетраоцетна киселина (EDTA) и 3 mM 2-меркаптоетанол) и съхранявани, както е описано в (32).
Същата стратегия беше приложена за получаване на контролните клетки. В този случай обаче индукторът винаги присъства в растежната среда, осигурявайки експресия на rplE в транс. Клетките бяха събрани при A600 ∼ 0.8–1.
Подготовка и анализ на рибозоми и рибозомни субединици
Рибозомите са получени съгласно (35) с модификации, публикувани в (32). За да се получат рибозомни субединици, рибозомите от изчерпаните с L5 клетки се зареждат върху 5–20% (тегло/тегло) градиенти на захароза, приготвени в буфер А (10 mM Tris – HCl, pH 7,5, 100 mM NH4Cl, 10 mM MgCl2 и 3 mM 2-меркаптоетанол) и се центрофугира при 64 000 g и 4 ° C в продължение на 11–12 h. При тези йонни условия непокътнатите 50S субединици остават свързани с 30S субединици, за да образуват 70S рибозоми, които значително улесняват пречистването на 45S частици. Рибозомните субединици от контролните клетки се приготвят по същия начин, но в присъствието на 1 mM MgCl2. Намаляването на концентрацията на магнезий до 1 mM и по-долу е конвенционален подход, позволяващ пълна дисоциация на 70S рибозоми върху 50S и 30S субединици (35). Отделените субединици се събират чрез високоскоростно центрофугиране, разтварят се в студен буфер А с 10% (обем/обем) глицерол, замразяват се и се съхраняват при -70 ° С. Големи рибозомни субединици от L5-изчерпани клетки бяха допълнително пречистени чрез второ центрофугиране в 5–20% (тегло/тегло) градиенти на захароза.
Афинитетно пречистване на 5S рРНК от цитоплазмата на L5-изчерпаните клетки
5S рРНК се изолира, използвайки афинитетна хроматография върху L5-Сефароза. Рибозомният протеин L5 се пречиства, както е описано по-рано (42) и ковалентно се прикрепя към Sepharose-4B съгласно (43). Без рибозома цитоплазматична фракция (S100) се инкубира с L5-Сефароза в буфер (10 mM Tris-HCl, pH 7.5, 50 mM NH4Cl, 10 mM MgCl2 и 3 mM 2-меркаптоетанол) при стайна температура за 30 минути. След това смолата се промива последователно със същия буфер, съдържащ 50 и 400 mM NH4CI. 5S рРНК и свързаните протеини се елуират с 3 М NaCl. Съдържанието на РНК и протеини в крайното измиване се анализира чрез електрофореза в денатуриращи условия, както е описано в (39) и (44), съответно. Количествата на протеиновите ленти са количествено използвани с помощта на софтуера ImageJ. Два протеина, притежаващи електрофоретична подвижност, подобна на тази на 5S rRNA-свързващи протеини L18 и L25, се елуират от гела и се идентифицират чрез масспектрометрия.
РЕЗУЛТАТИ И ДИСКУСИЯ
Растеж на изчерпаните с L5 клетки и образуването на рибозома
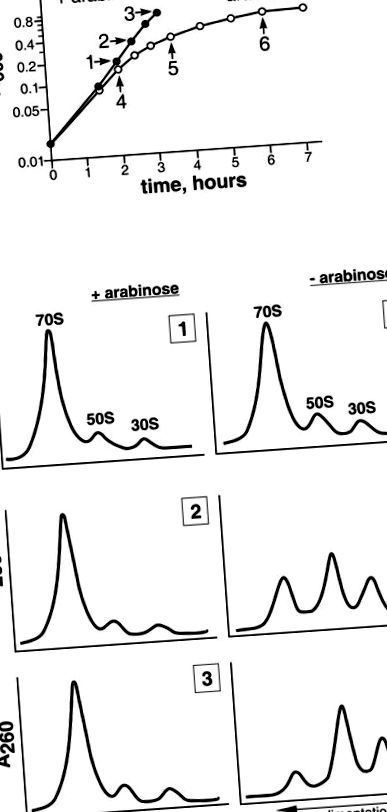
(A) Криви на растеж на клетките MS01 в средата с (запълнен кръг) или без (незапълнен кръг) индуктор. Стрелките показват времевите точки, когато са взети аликвотни части за анализ на рибозома. Цифрите (1–6) съответстват на рибозомните профили в раздел Б. (Б.) Седиментационен анализ на рибозоми от MS01, отглеждани или в присъствието (ляв панел), или в отсъствие (десен панел) на индуктора. Компонентите на клетъчната цитоплазма се фракционират чрез центрофугиране на 5–20% (тегло/тегло) градиент на захароза.
Свойства на големи рибозомни субединици и 5S рРНК-протеинов комплекс от L5-изчерпани клетки
Разпределение на 5S рРНК-протеинови комплексни компоненти в L5-изчерпани клетки. (A) Електрофоретичен анализ на РНК с ниско молекулно тегло (8% полиакриламиден гел, 8 М урея). 1—5S рРНК на Е. coli (контрол); 2 и 3 - S30 фракция от контролните и L5-изчерпани клетки, съответно; 4 и 5 - S100 фракция от контролните и L5-изчерпани клетки, съответно; 6 и 7 - големи рибозомни субединици от контролните и L5-изчерпани клетки, съответно. Пътища 2 и 3 съдържат сравними количества материал, както и платна 4 и 5. Пътека 7 е натоварена с два пъти повече материал от лента 6. (Б.) Протеини, които се пречистват с 5S рРНК чрез афинитетна хроматография върху L5-Сефароза (15% натриев додецил сулфат-полиакриламиден гел електрофореза). 1 — S100 фракция от изчерпаните с L5 клетки; 2—3 М солено измиване; 3 и 4 - Е. coli r-протеини L25 и L18, съответно.
Участие на протеин L5 в образуването на централната издатина на бактериалната рибозома
Протеинов състав на големи рибозомни субединици от контролата (A) и изчерпване на L5 (Б.) клетки чрез 2D гел електрофореза. Стрелките маркират позициите на r-протеините, които са обсъдени в текста. Геловете съдържат всички протеини на големи рибозомни субединици, с изключение на протеините L34 и L36. Протеин L34 е открит както в контролните, така и в 45S частиците при различни условия на разделяне. Протеин L36 не е анализиран.
(A) Модел на структурата на СР на рибозома на Е. coli. Позицията на протеина L31 се моделира в структурата на CP според позицията му в рибозомата на T. thermophilus. Посочени са позиции на рибозомни протеини, 5S рРНК и спирали на 23S рРНК. (Б.) Предложен модел за структурата на СР областта на бактериалната рибозома, образувана в отсъствието на протеин L5. Моделите са генерирани, използвайки кристалните структури на рибозомите на E. coli и T. thermophilus (PDB вписвания, съответно 2AW4 и 2J01). Изграждането на модели и усъвършенстването бяха извършени с помощта на програмата Coot; снимката е направена с помощта на PyMol.
Кооперативността и независимостта на сглобката CP предполага, че тя се появява като единна структурна единица не само по време на сглобяването на рибозома, но и при функционирането на рибозомата. Въз основа на междумолекулни кръстосани връзки в рибозомата, 5S рРНК беше предложено да бъде медиатор между няколко рибозомни функционални центъра (31). Според последните данни за структурна организация на рибозомите (9–13), компонентите на CP взаимодействат помежду си, като контактуват едновременно с повече от един компонент (Фигура 4 А). По този начин те образуват сложна автономна система в рамките на субединицата 50S, където промените в един компонент могат да бъдат предадени на останалите. Повечето от компонентите се свързват директно с 5S rRNP, което обяснява значението му за формирането на CP и предполага, че той играе ключова роля в CP функцията. В същото време СР компонентите контактуват директно с тРНК във всичките три рибозомни места (L16 и ASF в A-сайт, L5 в P-сайт и L33 в E-сайт); L27 изпъква в 50S субединицата, достигайки пептидилтрансферазния център; L25 се намира в непосредствена близост до свързания с GTPase център. С оглед на това предлагаме, че CP (като цяло) служи като координатор на функционирането на рибозомно активните центрове.
ДОПЪЛНИТЕЛНИ ДАННИ
Допълнителни данни са достъпни в NAR Online: Допълнителна таблица 1.
ФИНАНСИРАНЕ
Руската академия на науките, Руската фондация за фундаментални изследвания; програмата на RAS по молекулярна и клетъчна биология. Финансиране за такса за отворен достъп: Програма на RAS по молекулярна и клетъчна биология.
Изявление за конфликт на интереси. Никой не е деклариран.
- Свързана с полени хранителна алергия Липиден трансфер на протеини Сенсибилизация и корелация с
- Съображения за диети на растителна основа за въздействие върху околната среда, качество на протеините и ефективност на упражненията
- Информация за растителни протеини; Диаграма
- MuscleMeds Carnivor Телешки протеин Ягода - 56 порции
- Разклатете ги, това са най-добрите готови за пиене протеинови шейкове за отслабване уелнес капитан