Ибрахим Малами
1 Факултет по фармацевтични науки, Университет Усману Данфодийо, Сокото, Нигерия
Алию Мохамед
2 Катедра по биохимия, Факултет по науки за живота, Университет Ахмаду Бело, Зария, Нигерия
Имаобонг С. Ети
3 Катедра по фармакология и токсикология, Университет в Уйо, Уйо, Нигерия
Питър М. Уазири
4 Катедра по биохимия, Държавен университет Кадуна, Кадуна, Нигерия
Алхасан М. Алхасан
1 Факултет по фармацевтични науки, Университет Усману Данфодийо, Сокото, Нигерия
Резюме
Въведение
Материали и методи
Компютърен хардуер и софтуер
Симулацията на докинг протеин-лиганд се изпълнява на 4,00 GB RAM Intel® Core i5 2,5GHz с 64-битова операционна система Windows 10 (Acer Inc., New Taipei City, Тайван). 3D структурата на биоактивните съединения куркумин, флавокауаин В и алпинетиновото съединение, използвани за настоящото изследване, е изготвена с помощта на ChemOffice версия 5.1 (PerkinElmer, Waltham, MA, US) и е записана като PDB файл. Рентгеновата кристална структура на човешки R273H мутант p53 ядро домен протеин (2bim.pdb, 1.98 Å резолюция) (Joerger et al., 2006 [18]) и разтвор рентгенова кристална структура на разпознаване на ДНК в комплекс с диви- тип p53 (2ahi.pdb, разделителна способност 1.85 Å) (Kitayner et al., 2006 [20]) бяха извлечени от банката с данни за протеини (www.pdb.org) с помощта на визуализатора на Discovery Studio 4.5 (Accelryls, САЩ). Симулацията на докинг протеин-лиганд беше извършена с помощта на софтуерни инструменти Autodock версия 4.2 (Scripps Research Institute, US).
Изчислителни изследвания
(i) Докинг протеин-лиганд
Симулационното проучване за докинг протеин-лиганд първоначално се извършва с помощта на софтуерния инструмент Autodock. Водните молекули и други хетероатоми бяха напълно отстранени от кристалния протеин и всички липсващи водородни атоми бяха добавени към кристалния протеин. Файлът pdbqt на лиганда е приготвен чрез добавяне на заряди за гастеигър и отстраняване на неполярни водородни атоми към лигандите, докато техните заряди се сливат с техните въглеродни атоми. Корените на лигандите бяха открити и дефинираха техните въртящи се връзки, докато торсиите им бяха зададени на съответните им номера. Файлът с параметри на мрежата (gpf) беше изготвен чрез локализиране на мрежови карти при 60 × 60 × 60 Å в xyz решетъчни точки, 0,375 Å интервал и позицията на решетъчната кутия беше центрирана в остатъците от активното място около R248A и R273H мутантния протеин p53 с xyz-координати 98,591, 82,612 и 33,169 Å. Проведена е симулация на молекулярен докинг, като се използват енергийни оценки на 2 500 000 за брой от 100 GA пробега за всеки лиганд със 150 популационен размер. Генетичният алгоритъм на ламарския докинг е използван за идентифициране на местата на молекулярно свързване и прогнозиране на енергията на свързване на лиганд, константа на инхибиране, както и междумолекулна енергия (Morris et al., 2009 [29]). Подобен протокол беше извършен за всяка молекула, използвана в това изследване.
(ii) Докинг на протеин-нуклеинови киселини
Резултати
Оценка на взаимодействието протеин-лиганд
Молекулното докинг на протеин-лиганд се извършва, като се използва свободна енергия и силовото поле AMBER срещу кристалната структура на човешкия R273H мутант p53 ядро домен (Фигура 1 (Фиг. 1)). Впоследствие ние сме в състояние да предскажем свързването на куркумин, алпинетин и флавокауин В с целевия R273H мутант p53 протеин. Биоактивните съединения бяха успешно докирани срещу R273H мутантния протеин p53 с прогнозна дисоциационна константа съответно от 36,57, 70,77 и 75,11 цМ. Освен това енергията им на свободно свързване се изчислява съответно на -6,05, -5,66 и -5,63 kcal/mol за куркумин, флавокаван В и алпинетин. Крайната междумолекулярна енергия и общата вътрешна енергия са оценени на -9,04 и -1,62 kcal/mol за куркумин. От друга страна, се изчислява, че алпинетин и флавокауаин В се свързват с р53 при -6,52 и -0,43 и съответно -7,45 и -0,67 kcal/mol.
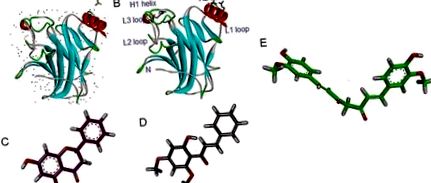
Различният режим на свързване на биоактивните съединения в повърхността на молекулярно свързване на R237H мутантния протеин p53 е показан на Фигура 3 (Фиг. 3). Съединенията са разположени заобиколени от страничните вериги на аминокиселините между L3 контура и H2 спирала на p53 протеин, съдържащ остатъци от ДНК-контакт.
Оценка на взаимодействието протеин-ДНК
Програма GRAMM и комбинация от QUASI-DNP, DFIRE и Varani потенциал бяха използвани за оценка на симулация на молекулно докинг на протеин-ДНК. Анализ на редокинг на разцепената кокристализирана структура на ДНК комплекс с див тип p53 първоначално беше извършен над сървъра NPDock, за да се тества точността и ефективността на използваното симулационно проучване за докинг-ДНК докинг. Следователно страничните вериги на ДНК остатъка от контакт Arg248 и Arg273 образуват директен контакт с ДНК фосфатния скелет. Едновременно с това се образува директен контакт от L3 контур през страничната верига Ser241 към ДНК фосфатния скелет. Освен това остатъкът Arg280 от H2 спиралата и остатъците от L1 веригата (Lys120 и Ser121) са свързани към гуаниновите основи (G8, G3 и G2) и ДНК фосфатния скелет (Фигура 4 (Фиг. 4)).
Разпознаването на контакт протеин-ДНК, получено от анализа на повторното опаковане, е практически идентично с това, получено от оригиналната съкристализирана структура (2ahi.pdb). Следователно, анализът на повторното опаковане потвърди валидността и ефективността на провеждащото се симулационно проучване на докинг протеин-ДНК на сървъра NPDock. Промените в резултатите на парцела, проверени по време на симулация на докинг на протеин-ДНК, са демонстрирани на Фигура 5 (Фиг. 5) .
Освен това се наблюдават съществени промени в структурата на свързваща повърхност и конформация на ДНК, включващи биоактивни молекули в сравнение с дивия тип р53-ДНК комплекс на Фигура 7 (Фиг. 7). Режимът на свързване на всяко от биоактивното съединение, наблюдавано в молекулярния свързващ джоб на мутантния р53-ДНК комплекс R237H, структурно изглежда идентичен. Режимът на свързване на мутант р53 към ДНК свързващата повърхност обаче се различава съществено между всички комплекси. В допълнение, леки промени в хидрофобността се наблюдават и в структурата на R273H мутант p53, включващ всички съединения.
- 7 възможни причини, поради които сте провалили мечтите, жени; s Здраве
- Мозъчни науки Безплатна пълнотекстова дълбока мозъчна стимулация - възможна стратегия за лечение при патология
- 9 възможни причини за отслабване на котки - Catster
- Функционален медицински подход към затлъстяването 10 начина за отслабване; Не го изключвайте - UltraWellness
- Нов подход за управление на гастропарезата