Роберто Канатаро
1 лаборатории GalaScreen, болница Mater Domini 88100 Катандзаро и Катедра по фармация, здравни и хранителни науки, Университет на Калабрия, 87036 Rende (CS), Италия
Мария Кристина Каролео
2 Катедра по фармация, здравни и хранителни науки, Университет на Калабрия, Via Pietro Bucci, 87036 Rende (CS), Италия
Алесия Фацио
2 Катедра по фармация, здравни и хранителни науки, Университет на Калабрия, Via Pietro Bucci, 87036 Rende (CS), Италия
Киара Ла Торе
2 Катедра по фармация, здравни и хранителни науки, Университет на Калабрия, Via Pietro Bucci, 87036 Rende (CS), Италия
Пиерлуиджи Пластина
2 Катедра по фармация, здравни и хранителни науки, Университет на Калабрия, Via Pietro Bucci, 87036 Rende (CS), Италия
Лука Галели
3 Оперативно звено по клинична фармакология и фармакологична бдителност, Катедра по здравни науки, Университет в Магна Греция, болница Mater Domini Catanzaro, 88100 Катандзаро, Италия
Graziantonio Lauria
2 Катедра по фармация, здравни и хранителни науки, Университет на Калабрия, Via Pietro Bucci, 87036 Rende (CS), Италия
Ерика Ционе
2 Катедра по фармация, здравни и хранителни науки, Университет на Калабрия, Via Pietro Bucci, 87036 Rende (CS), Италия
Резюме
Наскоро демонстрирахме способността на кетогенната диета (KD) да влияе на профила на експресия на микроРНК (miR). Тук съобщаваме, че KD е в състояние да нормализира miR експресията при затлъстели лица в сравнение с слаби субекти. Чрез прилагане на два различни инструмента за биоинформатика, ние открихме, че сред miRs, които се връщат на сравними нива при слаби субекти, четири от тях са свързани с антиоксидантни биохимични пътища, а другите са свързани както с антиоксидантни, така и с противовъзпалителни биохимични пътища. От особен интерес е регулирането на hsa-miR-30a-5p, което корелира с намаляването на протеина на експресия на каталаза в червените кръвни клетки.
1. Въведение
2. Материали и методи
2.1. Население
Етично одобрение за всички изследвания върху хора е дадено в съответствие с Регионалния комитет по етика (REC) (# 120-18052018). Субектите също са изключени, ако са показали наличие на хипертония и/или са били на лекарства. Смята се, че проучването не е организирало клинични изпитвания и не е регистрирано като такова. Писмено информирано съгласие е получено от участници, които отговарят на стандартите на етичната практика, както е посочено в декларацията от Хелзинки. Критериите за изключване включват диабет, бъбречни заболявания, чернодробна дисфункция, анамнеза за злоупотреба с алкохол или наркотици и неопластични заболявания през петте години преди проучването.
2.2. Имуноблот анализ и екстракция на РНК
Събират се кръвни проби и се събират червени кръвни клетки и лизати за каталазен (CAT) експресионен протеин, използвайки антитялото (abcam # ab16731). Концентрацията на протеин в пробите се изчислява, както е описано в [17,18,19,20]. След това се използва серумна плазма за анализ на функционална геномика. Общата РНК беше извлечена от 200 μL кръвен серум или плазма с помощта на miRNeasy Serum/Plasma Kit (Qiagen, Venlo, Холандия) с цел намаляване на потенциалните замърсители [15,21] и съгласно инструкциите на производителя.
2.3. Подготовка на проби от NanoString и анализ на данни
За n-броячът на NanoString Technology като вход се използва 100 ng RNA/miR. След това miRs бяха хибридизирани с nCounter Human-V3 miRNA експресионен анализ на CodeSet за една нощ при 65 ° C и както беше описано по-рано в [22,23]. За да се получат стабилни резултати, коефициентът на вариация (CV), изразяващ съотношението на стандартното отклонение към средната стойност и изразен като процент, беше избран като 30%. MiRs, за които е известно, че са свързани с кръвната хемолиза, бяха изключени от анализа [24,25].
2.4. In Silico Prediction of hsa-miR Target Genes
За да идентифицираме гените като мишени на hsa-miRs от масивния анализ, извършихме силико анализ. Идентификацията in silico на целевите гени беше извършена с помощта на miRTargetLink Human (https://ccb-web.cs.uni-saarland.de/mirtargetlink/) и DIANA Tools (http://diana.imis.athena-innovation.gr /DianaTools/index.php) бази данни. Тази последна база данни се използва за проверка кои миРНК целеви гени вече са валидирани експериментално.
2.5. Статистически анализ
маса 1
| Възраст, y | 46,5 ± 10,51 | 46,83 ± 12,32 | 46,6 ± 11,56 | ns | ||||
| Височина, см | 175,1 ± 5,2 | 171,3 ± 6,6 | 176,3 ± 3,3 | ns | ||||
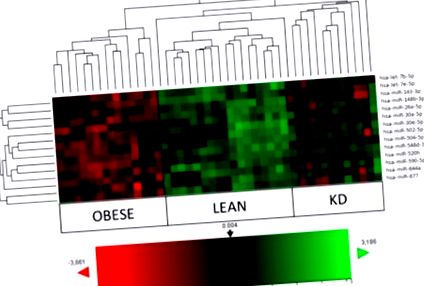
| Тегло, кг | 107,5 ± 3,0 | 70,8 ± 3,8 | 96,97 ± 11,2 | 2 | 33,9 ± 1,2 | 22,1 ± 2,5 | 31,5 ± 1,3 | Фигура 1, включително подписите на hsa-let-7b-5p, hsa-miR-143-3p, hsa-miR-148b-3p, hsa-miR-26a-5p, hsa-miR-502-5p, hsa-miR -520h, hsa-miR-548d-3p, hsa-miR-590-5p и hsa-miR-644a. По-специално, KD, в сравнение със субекти със затлъстяване, нормализира нивата на експресия на hsa-let-7b-5 (8/12), hsa-miR-143-3p (9/12), hsa-miR-148b-3p (10/12), hsa-miR-590-5p (10/12), hsa-miR-520h (8/12) и hsa-miR-644a (9/12), които бяха изразени при повече от 65% от пациентите, докато 100% беше постигнато за hsa-miR-548d-3p (12/12). Достигнати са поне 50% за hsa-miR-26a-5p (6/12) и hsa-miR-502-5p (7/12). Не се забелязва промяна за hsa-miR-504-5p. Тук идентифицираните нови hsa-miR-let7e-5p (5/12) и hsa-miR-877 (5/12) показват по-малко от 50% присъствие, с изключение на hsa-miR-30a-5p (8/12 ). |
Топлинна карта и йерархично групиране на субекти със затлъстяване (n = 14), слаби (n = 17) и кетогенна диета (KD) (n = 12) въз основа на най-различно експресираните микроРНК. Цветът и интензивността на кутиите представляват промени в генната експресия. В анализа червеното представлява гени, регулирани надолу, а зеленото - гени, регулирани нагоре. Черното представлява непроменен израз, както е видно от цветовата справка. Използван е софтуер n-Solver.
3.3. В Silico Резултати
За анализ in-silico бяха използвани две различни бази данни. Данните бяха сравнени по отношение на броя на целевите гени, валидирани експериментално в двете бази данни. Резултатите са представени в таблица 2. Въпреки че бяха намерени подобни резултати за два hsa-miRs (получер в Таблица 2), броят на потвърдените цели, намерени за други hsa-miRs, беше постоянно различен. В инструментите DIANA броят на валидираните целеви гени беше по-висок по отношение на miRtagertLink Human; следователно инструментите DIANA бяха използвани за по-нататъшен анализ на биоинформатиката.
Таблица 2
Инструменти за биоинформатика за in silico анализ.
| miRTargetLink Human | DIANA Инструменти | |
| hsa-let-7b-5p | 124 | 312 |
| hsa-let-7e-5p | 15 | 273 |
| hsa-miR-143-3p | 32 | 82 |
| hsa-miR-148b-3p | 10 | 218 |
| hsa-miR-26a-5p | 52 | 391 |
| hsa-miR-30a-5p | 119 | 458 |
| hsa-miR-30e-5p | 7 | 412 |
| hsa-miR-502-5p | 3 | 30 |
| hsa-miR-504-5p | 6 | 7 |
| hsa-miR-520h | 5 | 5 |
| hsa-miR-548d-3p | 1 | 203 |
| hsa-miR-590-5p | 2 | 43 |
| hsa-miR-644a | 2 | 0 |
| hsa-miR-877 | 0 | 19. |
3.4. Утвърдено hsa-miR взаимодействие и Western Blot анализ на каталаза
Прогнозираните и валидирани целеви гени бяха оценени с помощта на DIANA Tools. Установено е, че новият hsa-miR-let7e-5p регулира глутатион пероксидаза 7 (GPX7), както е показано чрез струнен анализ (Фигура 2 A), както и при взаимодействие на silico 3′UTR (Фигура 2 B). В silico 3′UTR области на тет метилцитозин диоксигеназа 3 (TET3) за hsa-miR-520h (Фигура 2 D) взаимодействие и 3′UTR области на супероксид дисмутаза 2 (SOD2) за hsa-miR-548d-3p (Фигура 2 F ) са показани, както и анализ на струни, съответно на Фигура 2 В, Д. Струнният анализ показа, че 10 протеина са способни да взаимодействат физически с GPX7 (Фигура 2 B), както и с TET3 (Фигура 2 D), SOD2 (Фигура 2 F) и каталаза (CAT) (Фигура 3 D). Този последен протеин е наблюдаван чрез Western blot анализ, както е показано на Фигура 3 А. Извършен е денситометричен анализ и е показано значително намаляване на нивата на CAT протеин, както е показано на Фигура 3 B. Установено е, че hsa-miR-30a-5p е насочен 3′UTR региони на CAT, както е показано на Фигура 3 C. Установено е обаче, че други целеви гени са повлияни от KD и свързани с антиоксидантния метаболизъм и свързаните с възпалението гени, както е показано в Таблица 3. Съкращения и имена на гени са описани в таблица 4 .
Биоинформатичен анализ. Взаимодействие с низови протеиниA) GPX7 и (Б.) GPX7 in silico взаимодействия с 3'UTR региона. Взаимодействие с низови протеини° С) TET3 и неговите in silico взаимодействия с 3'UTR региона в (д). Взаимодействие с низови протеини на (Е.) SOD2 и неговите in silico взаимодействия с 3′UTR региона в (F).
- Диета с ниско съдържание на мазнини, свързана с по-ниски нива на тестостерон при мъжете EurekAlert! Научни новини
- Диета с ниско съдържание на мазнини, свързана с по-ниско ниво на тестостерон Urology Times
- Еко спрей с висока ефективност Отслабване Отзиви Систематичен преглед Кетогенна диета Отслабване Conquip
- Опитах кетогенната диета в продължение на 30 дни
- Идентифициране на кетогенна диета за странични ефекти Фондация за епилепсия