Джордж Х. Пери
1 Училище за човешка еволюция и социални промени, Държавен университет в Аризона, Темпе, AZ 85287, САЩ
2 Отделение по патология, Brigham and Women’s Hospital, Бостън, Масачузетс 02115, САЩ
Натаниел Дж. Домини
3 Катедра по антропология, Калифорнийски университет, Санта Круз, Калифорния 95064, САЩ
Катрина Г. Нокът
1 Училище за човешка еволюция и социални промени, Държавен университет в Аризона, Темпе, AZ 85287, САЩ
4 School of Life Sciences, Arizona State University, Темпе, AZ 85287, САЩ
Артър С. Лий
2 Отделение по патология, Brigham and Women’s Hospital, Бостън, Масачузетс 02115, САЩ
Хайке Фиглер
5 The Welcome Trust Sanger Institute, The Wellcome Trust Genome Campus, Hinxton, Cambridge, CB10 1SA, Великобритания
Ричард Редон
5 The Welcome Trust Sanger Institute, The Wellcome Trust Genome Campus, Hinxton, Cambridge, CB10 1SA, Великобритания
Джон Вернер
4 School of Life Sciences, Arizona State University, Темпе, AZ 85287, САЩ
Фернандо А. Виланея
3 Катедра по антропология, Калифорнийски университет, Санта Круз, Калифорния 95064, САЩ
Джоана Л. Планина
6 Департамент по антропологични науки, Станфордски университет, Станфорд, Калифорния 94305, САЩ
Раджеев Мисра
4 School of Life Sciences, Arizona State University, Темпе, AZ 85287, САЩ
Найджъл П. Картър
5 The Welcome Trust Sanger Institute, The Wellcome Trust Genome Campus, Hinxton, Cambridge, CB10 1SA, Великобритания
Чарлз Лий
2 Отделение по патология, Brigham and Women’s Hospital, Бостън, Масачузетс 02115, САЩ
7 Харвардско медицинско училище, Бостън, Масачузетс 02115, САЩ
Ан С. Стоун
1 Училище за човешка еволюция и социални промени, Държавен университет в Аризона, Темпе, AZ 85287, САЩ
Свързани данни
Резюме
Консумацията на скорбяла е видна характеристика на земеделските общества и събирачите на ловци в сухи среди. За разлика от тях, тропическите гори и околоарктическите ловци и някои животновъди консумират много по-малко нишесте 1 - 3. Тази поведенческа промяна поражда възможността различни селективни налягания да са действали върху амилазата, ензима, отговорен за хидролизата на нишесте 4. Открихме, че броят на копията на гена на слюнчената амилаза (AMY1) корелира положително с нивата на слюнчените амилазни протеини и че индивидите от популациите с диети с високо съдържание на нишесте имат средно повече копия на AMY1 от тези с традиционно диети с ниско съдържание на нишесте. Сравненията с други локуси в подгрупа от тези популации предполагат, че нивото на диференциация на броя на копията на AMY1 е необичайно. Този пример за положителен подбор на гена с променлива номер на копие е един от първите в човешкия геном. По-високият брой на копията на AMY1 и нивата на протеини вероятно подобряват храносмилането на нишестените храни и могат да буферират срещу редуциращите фитнеса ефекти на чревни заболявания.
Еволюцията на хоминин се характеризира със значителни диетични промени, улеснени отчасти от развитието на технологията на каменните инструменти, контрола на огъня и наскоро опитомяването на растения и животни 5 - 7. Например, нишестето се превръща във все по-важен компонент от човешката диета, особено сред земеделските общества 8. Следователно има смисъл, че изследванията на еволюцията на амилаза при хората и близките ни роднини могат да предоставят представа за нашата екологична история. Тъй като човешкият ген на слюнчената амилаза (AMY1) показва значителни вариации в копие номер 9, 10, първо оценяваме дали съществува функционална връзка между броя на копията на AMY1 и нивото на експресия на амилазен протеин в слюнката. След това определяме дали броят на копията на AMY1 се различава сред съвременните човешки популации с контрастни нива на диетично нишесте.
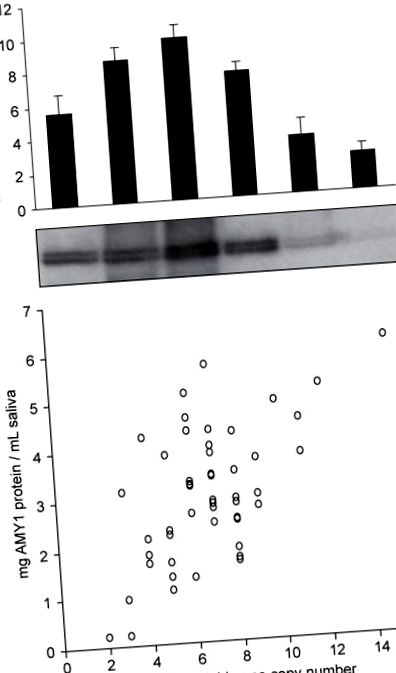
Вариация на номера на копие на AMY1 и експресия на слюнчен амилазен протеин. (а,б) От същите европейско-американски индивиди сме изчислили диплоиден номер на копие на ген AMY1 с qPCR (а) и нива на амилазен протеин в слюнката чрез Western blot (б). Лентите за грешки показват s.d. (° С) Връзка между диплоиден номер на копие на AMY1 и ниво на слюнчените амилазни протеини (n = 50 европейски американци). Значителна промяна в нивото на AMY1 протеин не се обяснява с броя на копията (R 2 = 0,351), което може да отразява други генетични влияния върху експресията на AMY1, като например единични нуклеотидни полиморфизми на регулаторната област (SNP) или негенетични фактори, които могат да включват индивидуални състояние на хидратация, ниво на стрес и краткосрочни хранителни навици.
Диета и вариация на броя копия на AMY1. (а) Сравнение на разпределението на честотата на диплоидно копие на броя на диплоидните копия AMY1 за qPCR за популации с традиционни диети, които включват много храни, богати на нишесте (високо нишесте) и популации с традиционни диети, които включват малко или никакво нишесте (ниско нишесте). (б) Кумулативен график на разпределение на диплоиден номер на копие на AMY1 за всяка от седемте популации в проучването.
Валидация на FISH с висока разделителна способност за оценка на броя копия на AMY1. Червените (∼10 kb) и зелените (∼8 kb) сонди обхващат целия AMY1 ген и ретротранспозон директно пред (и уникален за) AMY1, съответно. (а) Японският индивид GM18972 е оценен от qPCR, че има 14 (13,73 ± 0,93) диплоидни AMY1 генни копия, в съответствие с резултатите от фибри FISH, показващи единия алел с 10 копия, а другия с четири копия. (б) Индивидуалният GM10472 на Biaka е оценен от qPCR, че има 6 (6,11 ± 0,17) диплоидни AMY1 генни копия, в съответствие с резултатите от FISH на фибри. (° С) Референтното лице за шимпанзе (Clint; S006006) е потвърдено, че има две диплоидни копия на гена AMY1.
Схемите на популация на вариацията на броя на копията на AMY1 не отговарят на очакванията при прост регионален модел на генетичен дрейф: нашите проби с високо и ниско съдържание на нишесте включват африкански и азиатски популации, което предполага, че диетата по-силно предсказва броя на копията на AMY1, отколкото географска близост. Въз основа на това наблюдение предположихме, че естественият подбор може да е повлиял на броя на копията на AMY1 при определени човешки популации. Не можем обаче да проверим стриктно такава хипотеза само въз основа на нашите резултати от qPCR, отчасти защото липсват сравнителни данни от други локуси. Следователно ние извършихме сравнителна геномна хибридизация въз основа на масив (aCGH) върху пробата от популацията на якут с масивна платформа Whole Genome TilePath (WGTP), която преди това беше използвана от Redon и колеги 11 за описване на геномни модели на изменение на броя на копията в 270 индивиди (колекцията HapMap), включително същата японска популационна извадка, както в нашето проучване. За експериментите с якутски aCGH използвахме същата референтна ДНК проба (NA10851), както в предишното проучване 11, улеснявайки сравнението на японски и якутски съотношения на относителна интензивност log2 за 26 574 клона на бактериална изкуствена хромозома (BAC) върху масива, включително два клона картографиран в локуса AMY1.
Японско-якутска диференциация на номера на копие при AMY1 спрямо други локуси в целия геном. (а) Честотни разпределения на WGTP aCGH относителна интензивност log2 съотношения от AMY1-картографиран клон Chr1tp-6D2 за индивиди от Япония и Якут. (б) Връзката между японския и якутския среден коефициент log2 за всички автозомни WGTP клонинги, които са били променлива с номер на копие и в двете популации. AMY1-картираните клонинги Chr1tp-6D2 и Chr1tp-30C7 са изобразени съответно като плътни червени и сини кръгове.
В обобщение показахме, че моделът на вариация в броя на копията на човешкия ген AMY1 съответства на историята на свързания с диетата натиск за селекция, демонстрирайки значението на нишестените храни в човешката еволюция. Докато локусът на амилазата е един от най-променливите в човешкия геном по отношение на копие номер 10, той по никакъв начин не е уникален; скорошно проучване за целия геном идентифицира 1447 вариращи области на брой копия сред 270 фенотипично нормални човешки индивиди 11 и много повече такива региони вероятно ще бъдат открити с напредък в технологията за откриване на вариации на броя копия. Разумно е да се предположи, че варианти с брой копия, различни от AMY1, са или са били подложени на силен натиск от естествения подбор, особено като се има предвид тяхното потенциално влияние върху транскрипционните и транслационни нива (например, справка 29) Характеризирането на вариацията на броя на копията сред хората и между хората и другите примати обещава да даде значителна представа за нашата еволюционна история.
Методи
Проби
Букални тампони и слюнка бяха събрани при информирано съгласие от 50 европейски американци на възраст 18-30 години (протокол IRB на държавния университет в Аризона № 0503002355). Слюнката се събира за 3 минути под езика. Букални тампони бяха взети от Hadza (n = 38) и Datog (n = 17) от Танзания (IRB протокол № 9798-414 на Станфордския университет). Геномните ДНК от Биака (Централноафриканска република; n = 32), Mbuti (Демократична република Конго; n = 15) и Якут (Сибир; n = 25) са от клетъчната линия за разнообразие от човешки геном HGDP-CEPH. Лимфобластоидни клетъчни линии от 45 японски, 4 допълнителни Biaka и донора за геномната последователност на шимпанзето (Clint) са получени от Института за медицински изследвания на Coriell. По време на рутинните ветеринарни прегледи от шимпанзета и бонобо, настанени в различни зоологически и изследователски съоръжения, бяха събрани цели кръв. Две допълнителни проби от бонобо са получени от Интегрираните биоматериали за примати и информационен ресурс. ДНК се изолира с помощта на стандартни методи.
Копиране на оценка на номера
Праймерите за qPCR (допълнителна таблица 3 онлайн) са проектирани да бъдат специфични за AMY1 (т.е. несъответствия на последователности с AMY2A и AMY2B) на базата на референтни геномни последователности на човек и шимпанзе. Предишно проучване съобщава за единично (хаплоидно) копие на AMY1 за едно шимпанзе 18 и скорошен анализ от Cheng et al. 19 не откриха доказателства за скорошно дублиране на AMY1 за Clint. Използвахме фибри FISH, за да потвърдим, че Clint има две диплоидни копия на AMY1 (фиг. 3в). Следователно успяхме да изчислим броя на диплоидното копие въз основа на относително количество AMY1 за човешки ДНК в сравнение със стандартна крива, изградена от ДНК на Clint. Фрагмент от гена TP53 също беше амплифициран, за да се коригира вариацията на количеството на разредено ДНК. Пробите бяха изготвени в три екземпляра, а стандартите в два екземпляра. Експериментите бяха проведени и анализирани, както е описано 20 .
Western blot анализ
Протеиновите проби се приготвят чрез разтваряне на проби от слюнка в 2% натриев додецил сулфат (SDS) и нагряване при 100 ° С в продължение на 5 минути. Тези проби бяха анализирани върху мини SDS-полиакриламидни гелове и прехвърлени в мембрани от поливинилиден дифлуорид (PVDF) (Immobilon-Millipore). За целите на количественото определяне, върху всеки гел се пуска проба от човешки слюнчен амилазен протеин с известно количество (Sigma), с 5 μL слюнка за всяка проба. След прехвърляне мембраните се инкубират в продължение на 1,5 часа с първични антитела, издигнати срещу човешка слюнна амилаза (Sigma). Мембраните се промиват и се добавят конюгирани IgG кози анти-заешка алкална фосфатаза вторични антитела (Pierce) за 1 час. Мембраните бяха изложени на ECF субстрат (Amersham Biosciences) в продължение на 5 минути и анализирани с помощта на фосфоример. Количественото определяне на протеиновите ленти беше извършено с помощта на софтуера ImageQuant (Molecular Dynamics).
Фибри FISH
ДНК влакната се приготвят чрез внимателно лизиране на култивирани лимфобластни клетки с 300 μl буфер за лизис на клетки (Gentra Systems) на 5 милиона клетки. 10 μl лизат се поставя върху предметно стъкло с покритие от поли-L-лизин (LabSciaching) и се разтяга механично с ръба на покриващо стъкло. След 30 секунди се прилагат 300 μl 100% метанол за фиксиране на влакната. Слайдовете се изсушават при 37 ° С в продължение на 5 минути и след това се съхраняват при стайна температура (RT).
PCR продуктови сонди са направени от (i) целия AMY1 ген (~ 10 kb; червен на изображения) и (ii) ретротранспозона, намерен директно над всички копия на AMY1, но не и гени на амилаза на панкреаса или псевдогени на амилаза (~ 8 kb; зелено в изображения); докато генната сонда може да не е специфична за AMY1 при всички условия на хибридизация (дивергенция на AMY1 последователност с AMY2A и AMY2B = съответно 7,5% и 7,1%), пробата нагоре по веригата е. Използвахме дълги разстояния, последвани от вложени PCR за всеки регион (праймери и условия са предоставени в Допълнителна таблица 3 онлайн). PCR продуктите се пречистват с DNA Clean и Concentrator колони (Zymo).
За всеки вложен PCR продукт, 750 ng се комбинират с 20 μl 2.5x случаен праймер (BioPrime aCGH етикетиращ модул, Invitrogen) в общ обем 39 μl, поставен при 100 ° C за 5 минути и след това за 5 минути лед. След това 5 μl 10x dUTP и 1 μl Exo-Klenow фрагмент (BioPrime модул) и 5 μl (5 nmol) Biotin-16-dUTP (Roche; генна сонда) или 5 μl (5 nmol) дигоксигенин-11-dUTP ( Roche; сонда нагоре по веригата) бяха добавени и инкубирани при 37 ° С в продължение на 5 часа. Етикетираните продукти се пречистват с центробежни филтърни устройства Microcon (Millipore), като се използват 3 промивки от 300 μl 0,1x SSC, елуирани с 50 μl H2O. За всеки 1 μg маркирана ДНК, ние добавихме 10 μg човешка Cot-1 DNA (Invitrogen).
За всеки експеримент 500 ng маркирана ДНК от всяка от вложените PCR реакции се комбинират, лиофилизират, възстановяват в 10 μl хибридизационен буфер (50% формамид, 20% декстран сулфат, 2x SSC) и се добавят към предметното стъкло (18 × 18 mm покриващо стъкло; Fisher). Влакната и сондите бяха ко-денатурирани (95 ° С за 3 минути) и хибридизирани в овлажнена камера (37 ° С за 40 часа). Плъзгачът се измива в 0.5x SSC при 75 ° С в продължение на 5 минути, последвано от 3 измивания в 1x PBS при RT за 2 минути всяко. След това влакната се инкубират с 200 μl CAS блок (Zymed) и 10% нормален кози серум (Zymed) в продължение на 20 минути при RT под HybriSlip (Invitrogen). Използвахме 3-стъпково откриване/усилване (с реагенти в 200 μl CAS блок/10% нормален кози серум). Всяка стъпка беше 30 минути при RT под HybriSlip, последвана от 3 измивания в 1x PBS за по 2 минути при RT: (i) 1: 500 Анти-дигоксигенин-флуоресцеин, Fab фрагменти (Roche) и 1: 500 Strepavidin, Alexa Fluor 594 конюгат (Invitrogen); (ii) 1: 250 заешки анти-FITC антитела (Zymed) и 1: 500 биотинилиран анти-стрептавидин (Vector Laboratories); (iii) 1: 100 кози анти-заешки IgG-FITC (Zymed) и 1: 500 стрепавидин, конюгат Alexa Fluor 594. Изображенията са заснети на флуоресцентен микроскоп на Olympus BX51 с камера Applied Imaging и са анализирани със софтуера Genus на Applied Imaging.
aCGH анализ
За експерименти с aCGH използвахме клониран ДНК микрочип с голяма вложка, покриващ човешкия геном при разделителна способност на трасето 30. Тестовите (индивиди от Якут) и референтните (NA10851) геномни ДНК проби бяха маркирани съответно с Cy3-dCTP и Cy5-dCTP (NEN Life Science Products) и кохибридизирани с масива. За всяка проба беше извършен дублиран експеримент при размяна на багрило, за да се намалят честотите на грешно положителни грешки. Етикетирането, хибридизацията, измиванията и анализите се извършват, както е описано на 11, 30 .
- DZ Ep 22 CJ Hunt, Perfect Human Diet - The Disrupt Zone
- Есе за здравословна диета Топ 7 есета Човешки същества Биология
- Циклична кето диета Всичко, което трябва да знаете за тази разновидност на кетогенната диета PINKVILLA
- Еволюция на диетата в животинското дърво на живота - Román ‐ Palacios - 2019 - Evolution Letters -
- Диета и генна експресия Вие сте това, което ядете Станфорд онлайн