Xuemei Yuan
Департамент по биологични науки и Център за структурна биология, Имперски колеж по наука, технологии и медицина, 1 лаборатория Wolfson, Exhibition Road, London SW7 2AY, UK и 2 Blackett Laboratory, Prince Consort Road, London SW7 2BW, UK
Наталия Давидова
Департамент по биологични науки и Център за структурна биология, Имперски колеж по наука, технологии и медицина, 1 лаборатория Wolfson, Exhibition Road, London SW7 2AY, UK и 2 Blackett Laboratory, Prince Consort Road, London SW7 2BW, UK
Стивън Къри
Департамент по биологични науки и Център за структурна биология, Имперски колеж по наука, технологии и медицина, 1 лаборатория Wolfson, Exhibition Road, Лондон SW7 2AY, Великобритания и 2 лаборатория Blackett, Prince Consort Road, Лондон SW7 2BW, Великобритания
Стивън Матюс
Департамент по биологични науки и Център за структурна биология, Имперски колеж по наука, технологии и медицина, 1 лаборатория Wolfson, Exhibition Road, London SW7 2AY, UK и 2 Blackett Laboratory, Prince Consort Road, London SW7 2BW, UK
Резюме
Полипиримидиновият тракт, свързващ протеин (PTB), хомодимер, който съдържа четири RRM тип РНК свързващи домена на мономер, играе важна роля както в регулирането на алтернативното сплайсинг, така и в стимулирането на инициирането на транслация, както е указано от вътрешните места за влизане на рибозоми на някои пикорнавируси . Използвали сме експерименти за картографиране на химически отмествания, за да изследваме взаимодействията между PTB-34, рекомбинантен фрагмент, който съдържа третия и четвъртия RRM домейни на протеина, и редица къси богати на пиримидин РНК олигонуклеотиди. Резултатите потвърждават, че РНК взаимодействат предимно с повърхността на β-листа на PTB-34, но също така разкриват ролята на двата дълги гъвкави линкера в протеиновия фрагмент, резултат, който се подкрепя от експерименти с мутагенеза. Картирането показва различни предпочитания за свързване за RRM3 и RRM4, като първият прави особено специфично взаимодействие с последователността UCUUC.
ВЪВЕДЕНИЕ
Полипиримидиновият тракт, свързващ протеин (PTB), е РНК свързващ протеин, който функционира, за да регулира използването на пратеника и преди съобщението РНК в редица различни контексти. Докато основната физиологична роля на PTB изглежда е специфична за тъканите регулация на алтернативно сплайсиране на голям брой гени, като α-актинин, α- и β-тропомиозин, c-src, рецептори на растежен фактор на фибробластите и γ2 субединицата на рецепторът GABAA (1-11), протеинът също е замесен в контрола на полиаденилирането (12,13) и локализацията на иРНК (14). Освен това, PTB се набира от редица пикорнавируси като стимулатор на иницииране на транслация, задвижван от вътрешни места за влизане на рибозоми (IRES) (15–20). Неотдавнашна работа сочи, че протеинът може също да регулира транслацията на вируса на хепатит С, като взаимодейства както с 5 ′, така и с 3 ′ края на вирусната РНК (21,22) и че може да стимулира активността на клетъчните IRES на APAF-1 (23).
PTB функционира във всички тези системи чрез взаимодействие с РНК и други спомагателни протеини (фактори за сплайсинг и иницииране на транслация), въпреки че механизмите на действие остават неясни. Наблюдава се, че местата за свързване на PTB в интрон и IRES РНК съдържат повторения на кратки пиримидинови мотиви (напр. UCUU, UCUUC, UUCUCU, CUCUCU) (1,3,8,11,24), които често, но не изключително, се съдържат в богат на пиримидин фон. Тези наблюдения се подкрепят от резултати от in vitro селекция (8,25) и експерименти със свързване (26).
PTB е хомодимер (26) и съдържа четири RRM тип RNA свързващи домейни на мономер (27). Изследванията на делеционна мутагенеза са картографирали първичната РНК свързваща активност към третия и четвъртия RRM домейни на всеки мономер, въпреки че приносът на RRM1 и RRM2 също може да бъде значителен (17,26,28-30). RRM2 изглежда играе важна роля в димеризацията на PTB (26,28). Структурата на разтвора на PTB-34, мономерен фрагмент, който съдържа третия и четвъртия RRM домейни на PTB, разкрива, че RRM3 има нетипична структура за този тип модул, като четиривитовия β-лист, който действа като RNA свързване повърхността е удължена с една нишка (29). Структурата на PTB-34 също е доминирана от два разширени и гъвкави линкерни полипептида, единият (17 аминокиселини), свързващ β-веригите 4 и 5 на RRM3, а другият (25 аминокиселини), свързващ двата RRM домена, което показва, че протеинът има висока степен на конформационна изменчивост, поне преди взаимодействието си с РНК.
За да разширим нашите структурни изследвания на взаимодействията на PTB с РНК мишени, ние извършихме експерименти с картографиране на химически смени върху комплекси между PTB-34 и редица къси, синтетични, богати на пиримидин РНК молекули. Този експериментален подход разкрива аминокиселините, които са в контакт с РНК лиганда. Сравнението на различното разпределение на аминокиселинните контакти, наблюдавани с различни РНК олигонуклеотиди, разкрива нови прозрения относно спецификата на последователността на PTB.
МАТЕРИАЛИ И МЕТОДИ
ЯМР спектроскопия
15 N-маркиран PTB-34, който съдържа остатъци 335–531 от човешки PTB-1, се експресира в Escherichia coli и се пречиства, както е описано по-горе (29). Синтетичните РНК олигонуклеотиди бяха приготвени и пречистени с гел от Dharmacon Research, Inc. За експерименти с NMR картографиране, РНК олигонуклеотидите бяха титрувани в разтвори, съдържащи 0.5 mM 15 N-белязан PTB-34 в 20 mM натриев ацетат, рН 5.4. 15 N- 1 H HSQC спектри (31) бяха записани при RNA: PTB-34 молни съотношения от 0,5: 1, 1: 1 и 2: 1, за да се улесни проследяването и по този начин определяне на резонанси, нарушени от свързването на РНК с протеина. Всички ЯМР спектри бяха получени при 302 К, използвайки четириканален Bruker DRX500, оборудван с z-екраниран градиент и тройна резонансна сонда. NMR данните бяха обработени с помощта на NMRPipe/NMRDraw (32) и анализирани с помощта на NMRView (33).
Мутагенеза и експерименти със свързване с РНК
Мутантите на hisPTB-34a (остатъци 324–531 от човешки PTB-1) се приготвят чрез припокриване на PCR, като се използват установени протоколи и се експресират в E.coli (29). Всички мутации бяха потвърдени чрез cDNA секвениране. Експериментите за свързване на PTB-RNA бяха извършени, използвайки α- 32 Р-белязани РНК транскрипти на домейн 1 от EMCV IRES (17) в тестове за свързване на нитроцелулозен филтър, както е описано (29). Накратко, реакциите на свързване на РТВ-РНК (75 µl) бяха приготвени и инкубирани за 24 ° C поне 15 минути в 10 mM HEPES (рН 7.25), 100 mM KCl, 3 mM MgCl2, 5% глицерол, 1 mM DTT, 50 µg/ml дрожди tRNA (Boehringer Mannheim), 50 µg/ml човешки серумен албумин (Delta Biotechnology). Концентрацията на РНК обикновено е фиксирана на 4 nM. Тестовете бяха проведени, използвайки нитроцелулозна мембрана Protran BA-85, свързваща протеини (Schleicher и Schuell). Мембраната се измива обилно в 10 mM HEPES (рН 7.25), 3 mM MgCl2, 5% глицерол, 1 mM DTT и се монтира върху 96-гнезден блок за измерване на точки (Bio-Rad). Преди и след нанасяне на 65 ul от реакцията на свързване, мембраната се измива с 180 ul промивен буфер. След експеримента мембраната беше изсушена и количеството на свързания PTB-RNA комплекс беше определено чрез сцинтилационно броене на радиацията на Черенков. Освен ако не е посочено друго, всички реагенти са закупени от Sigma-Aldrich.
РЕЗУЛТАТИ
Селекция на РНК олигонуклеотиди
Естествените PTB свързващи сайтове се състоят от множество повторения на къси пиримидинови последователности, вградени в участък от интрон или IRES РНК, който обикновено е с дължина> 100 nt. Такива големи цели не могат лесно да бъдат анализирани от ЯМР, така че нашите изследвания се фокусираха върху свързването на кратки мотиви. Избраните последователности варират с дължина от 4 до 10 nt и включват мотиви, идентифицирани преди това в местата за свързване на PTB или леки вариации на тези (Таблица (Таблица 1 1).
маса 1.
| 1 | UCUU а | Каспаза-9 (4); клатринова лека верига B (11); пептид, свързан с калцитонин/калцитониращ ген (13); рецептор за растеж на фибробласти-1 (51); GABA y2 субединица (1); N-метил-d-аспартатен рецептор (11); а-актинин (5); а-тропомиозин (8); β-тропомиозин (6) |
| 2 | CUUC | |
| 3 | UCUU° С | |
| 4 | UCUUU a | Рецептор за растеж на фибробласти-2 (2) |
| 5 | UCUCU | Каспаза-9 (4); клатринова лека верига B (11); c-src (52); GABA y2 субединица (1); а-тропомиозин (8); β-тропомиозин (6) |
| 6 | C UCUCU | c-src (52); α-тропомиозин (8) |
| 7 | UCCUCUU° С | Както по-горе за UCUUC |
| 8 | UCUU CUCU | b Каспаза-9 (4); клатринова лека верига B (11); c-src (52); GABA y2 субединица (1); а-тропомиозин (8); β-тропомиозин (6) |
| 9 | UCUU ° С UCUU° С |
UCUU последователности, подчертани с удебелен шрифт; Подчертани последователности на UCUCU.
a Мотиви, идентифицирани в сайтове, защитени от PTB от химическа модификация и разцепване на RNase T1 в EMCV и FMDV IRES последователности (24,44).
b Позоваванията са на появата на хексамерния мотив UUCUCU.
Картиране на мястото на свързване на РНК на PTB-34
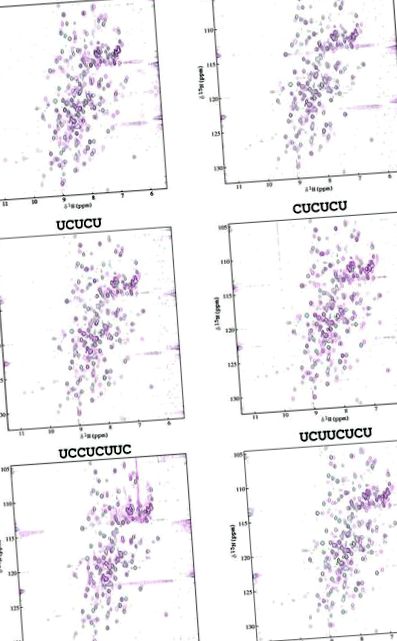
Фигура Фигура 1 1 показва наслагвания на HSQC спектри на равномерно 15 N-маркиран PTB-34 с и без еднакво моларно количество лиганд РНК за олигонуклеотиди 3–8. Трябва да се отбележи, че няколко резонанса, принадлежащи към силно гъвкавия контур, свързващ β-вериги 4 и 5 в RRM3 или дългия гъвкав междудоменен линкер, не могат да бъдат присвоени в апо-протеина поради тежко припокриване в центъра на спектъра (29 ). Много от тях показват промени в химичното изместване при свързване с РНК, но не беше възможно да се запишат триизмерни NMR данни за комплексите (за да се улесни специфичното определяне на последователността), тъй като РНК олигонуклеотидите предизвикаха разграждане на протеиновата проба в рамките на 8 часа.
Наслояване на 15 N- 1 H HSQC спектри на 15 N-PTB-34 ± РНК, записани при рН 5,38, 302 К. Спектрите на apo-PTB-34 са показани в черно, а тези на holo-PTB-34 в магента.
Комбинирано смущение с химично изместване (Δδ) на скелета 15 N и 1 HN резонанси на PTB-34 при свързване на РНК. Абсолютните стойности на 15 N и 1 HN промени в химичното изместване се комбинират, като се използва това уравнение: Δδ (15 N + 1 HN) = │Δδ 15 N│/4.69 + │Δδ 1 HN│. Корекционният коефициент за 15 N промени в химичното изместване се изчислява като съотношението на спектралната ширина на 15 N спрямо това на 1 Н (46,47). 0,1 pppm прагът е обозначен с хоризонтална линия.
Преглед на аминокиселинните остатъци в PTB-34 със значителни промени в химичните измествания при свързване на РНК. Остатъци с комбинирано изместване на възмущението от ≥0,1 p.p.m. за поне четири от шестте РНК олигонуклеотиди, които се свързват с протеина, са засенчени в зелено или оранжево. Зеленото засенчване показва, че страничната верига на аминокиселината е изложена на разтворител; заровените странични вериги са засенчени в оранжево. Хексамерните RNP-2 и октамерните RNP-1 мотиви и в двата RRM са в кутия. Звездичките маркират позициите на остатъци, които са ароматни в повечето RRM последователности (48). Мутация на остатъци, маркирани с ‘(Фиг.3). 3). PTB се отличава с отсъствието на триада ароматни остатъци, която се намира в повечето други RRM протеини и прави важни взаимодействия при подреждане с RNA основи. Установено е обаче, че някои от хидрофобните остатъци, които в PTB заемат тези общо запазени ароматни позиции (Leu 340 и Leu 378 в RRM3, Met 493 в RRM4), имат значителни химически промени в изместването, което показва, че те поддържат роля в свързването на РНК. Тези алифатни странични вериги в PTB могат да осъществяват подобни взаимодействия с хидрофобните Ile-base контакти, наблюдавани в кристалните структури на секс-летален и HuD (35,36). Въпреки че трите останали „ароматни“ позиции в PTB (Asn 376 в RRM3, His 457 и Leu 495 в RRM4) показват само малки или незначителни промени в химичното изместване в присъствието на РНК, това не изключва участието им в РНК взаимодействия. Всъщност се наблюдава мутация на His 457 за намаляване на свързването с РНК (29).
Сравнението на моделите на химични измествания, свързани с различните РНК олигонуклеотиди, разкрива редица интригуващи разлики. Последователността UCUUC предизвиква химически отмествания, които са почти изключително групирани върху свързващата повърхност на RRM3, което показва, че този олигонуклеотид може да бъде специфично свързан с RRM3 (фиг. (Фиг. 4). 4). Това тълкуване се подкрепя от наблюдението, че РНК-деканмерът (UCUUCUCUUC, олигонуклеотид 9), съдържащ две копия на UCUUC последователността, дава значително разширяване на линията в целия спектър (данните не са показани), показателни за образуването на високомолекулен тегловен комплекс, вероятно с протеин: РНК стехиометрия по-голяма от 1: 1. Едно просто обяснение за това би било, че две молекули PTB-34 се свързват с олигонуклеотида предимно чрез техните RRM3 модули. Олигонуклеотидът UCUUCUCU, който се различава от олигонуклеотид 9 само по това, че му липсват 2 nt в 3 ′ края, не причинява същата димеризация, засилвайки извода, че са необходими две копия на UCUUC за свързването на повече от една молекула PTB-34 с олигонуклеотидът.
Незначителни модификации на последователността на UCUUC, за да се получат UCUUU и UCUCU, драстично променят разпределението на химичните отмествания, като се получава по-голяма повърхност на смущение, която се простира, за да покрие голяма част от повърхността на β-листа в RRM4 (фиг. (Фиг. 4). 4 ). Кристалната структура на N-крайната двойка RRM домейни на поли (А)-свързващ протеин (PABP), комплексиран с поли (А), разкрива, че РНК се свързва в силно разширена конфигурация, така че 6 nt са достатъчни, за да обхванат RRM двойката (37). UCUUU и UCUCU пентамерите могат да приемат подобни разширени конфигурации, когато са свързани с PTB-34, интерпретация, подкрепена от наблюдението, че промените в химичното изместване са били близки до максималните при молно съотношение 1: 1 РНК: PTB-34. Независимо от това, възможно е разпределените модели на отмествания за тези два олигонуклеотида да се дължат на преместването на лигандите през повърхностите на β-листа в рамките на двата RRM домена, така че наблюдаваната повърхност на смущение представлява средна стойност на различни свързващи конформации. Модел на отмествания, който се простира през RRM3 и RRM4, също е наблюдаван за хексамера (CUCUCU), който показва много подобна повърхност на смущение до тази на UCUCU (виж Фигури Фиг. 1 1 и и 4). 4). Изглежда, че допълнителният пиримидин (цитозин) в 5 ′ края не е повлиял специфичността на свързване.
Двата октамера UCCUCUUC и UCUUCUCU съдържат UCUUC мотив, но имат три допълнителни пиримидинови остатъка съответно на 5 'и 3' края. Мащабът на химичните смущения на изместване за UCCUCUUC са значително по-малки, отколкото за UCUUC (Фиг. (Фиг. 2), 2), което означава, че UCCUCUUC предизвиква по-малки конформационни промени в протеина или причинява допълнително усредняване на химичното изместване за няколко конформации. По този начин, специфичността на свързване на последователността UCUUC за RRM3 изглежда е била нарушена в UCCUCUUC. За другия октамер UCUUCUCU, степента на конформационно смущение до RRM3 е подобна на тази за UCUUC; но в допълнение, той също причинява значителни промени в RRM4 със сходна величина и специфичност на тази на UCUCU пентамера (Фиг. (Фиг. 4), 4), който съответства на петте остатъка в 3 ′ края на UCUUCUCU.
ДИСКУСИЯ
ПРИЗНАВАНИЯ
Благодарим на Филип Шарп (MIT, САЩ), Ричард Джаксън и Ан Камински (Университет Кеймбридж, Великобритания), Греъм Белшам (IAH, Пирбрайт, Великобритания) и Дъг Блек (UCLA, САЩ) за реагентите и дискусиите. Тази работа се финансира от безвъзмездна помощ от Trust Trust и Биотехнологични и биологични науки, Великобритания.
- Химическо картографиране на РНК взаимодействията с полипиримидиновия тракт, свързващ протеин Nucleic
- FlaF е β-сандвич протеин, който закрепва архаела в обвивката на археалната клетка чрез свързване
- Разберете дали високо протеиновите диети и косопадът са свързани коса за коса
- Златни протеинови хапки
- Употреба на Goldthread, странични ефекти, взаимодействия, дозировка и предупреждение