Свързани данни
Резюме
Микробно разнообразие на здрави хора
Метагеномичните четения от 127 проби от език, обхващащи 90 субекта, са обработени с MetaPhlAn, за да се определят относителните количества в отделните видове. А) Относително изобилие от 11 различни Streptococcus spp. В допълнение към вариациите в по-широките кори (виж фиг. 2), отделните видове в рамките на едно местообитание демонстрират широк спектър от композиционни вариации. Вложката илюстрира средния състав на пробата на езика. Б) Метаболитни модули, присъстващи/отсъстващи (сиви/бели) в KEGG 28 референтни геноми на стрептококи на езика означават избрани области на специфична за щама функционална диференциация. В) Сравнително геномно покритие за единичния щам Streptococcus mitis B6. Сивите точки са средно отчитане на килобаза на милион четения (RPKM) за прозорци 1kb, сивите ленти са 25-ия до 75-ия персентил във всички проби, червената линия на изгладената средна стойност за ниски нива. Червените ленти в долната част подчертават предсказаните геномни острови 31. Големите, дискретни и силно променливи острови обикновено са недостатъчно представени. Г) Осветени два острова, V = V-тип H + ATPase субединици I, K, E, C, F, A & B и CH = холин-свързващи протеини cbp6 и cbp12, което показва функционална кохезия на специфична за щама генна загуба в рамките на отделен човек домакини.
Не са наблюдавани таксони, които да присъстват универсално сред всички телесни местообитания и индивиди на използваната тук дълбочина на секвениране, за разлика от няколко пътеки (фиг. 2 и Supp. Фиг. 2, виж по-долу), въпреки че няколко клада демонстрират широко разпространение и относително изобилие от модели на пренасяне 7–9. Вместо това, както се предлага от индивидуално фокусирани проучвания 3–4,6,10–11, всяко местообитание на тялото в почти всеки субект се характеризира с един или няколко таксони с подпис, съставляващи множеството на общността (фиг. 3). Сигнатурните класове на ниво род се формират средно от 17% до 84% от съответните им местообитания, напълно отсъстващи в някои общности (0% при това ниво на откриване) и представляващи цялата популация (100%) в други. Поразително е, че по-малко доминиращите таксони също бяха силно персонализирани, както сред индивидите, така и сред телесните местообитания; например в устната кухина повечето местообитания са доминирани от Streptococcus, но те са последвани в изобилие от Haemophilus в устната лигавица, Actinomyces в наддесневата плака и Prevotella в непосредствено съседната (но с ниско съдържание на кислород) субгингивална плака 12 .
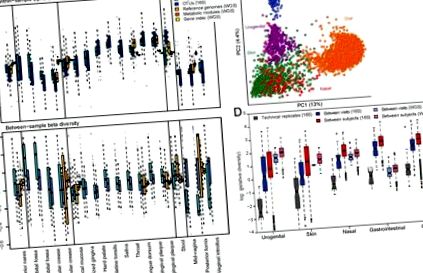
Вертикалните ленти представляват проби от микробиоми по местообитания на тялото в седемте местоположения с данни както за пушка, така и за 16S данни; стълбовете показват относително изобилие, оцветено от A) микробна фила от смесени OTU и B) метаболитни модули. Легендата показва средно най-разпространените фила/пътища в едно или повече телесни местообитания; RC = ретроаурикуларна гънка. Множество членове на повечето общности се състоят от един доминиращ тип (и често род; вж. Доп. Фиг. 2), но това е универсално нито за всички местообитания на тялото, нито за всички индивиди. И обратно, повечето метаболитни пътища са равномерно разпределени и разпространени както в индивидите, така и в телесните местообитания.
A – C) Разпространение (интензивност, цвят, обозначаващ тип/клас) и изобилие, когато присъстват (размер) на кладовете в здравия микробиом. Най-разпространените A) метагеномично идентифицирани видове, B) 16S-идентифицирани родове и C) PATRIC 14 „патогени“ (метагеномни). Размерът на популацията и дълбочините на секвениране на HMP са дефинирали добре микробиома на всички анализирани места на тялото, както се оценява чрез насищане на добавена общност D) метаболитни конфигурации (разреждане на минималното β-разнообразие на Bray-Curtis от метагеномни изобилия на ЕС до най-близкия съсед, междуквартилен обхват над 100 проби) и E) филогенетични конфигурации (мин. 16S OTU претеглено разстояние UniFrac до най-близкия съсед).
Превоз на специфични микроби
Метаболизъм и функция на микробиома
Като първото проучване, което включва както маркерен ген, така и метагеномни данни за телесни местообитания от голяма човешка популация, ние допълнително оценихме екологията на микробните метаболитни и функционални пътища в тези общности. Реконструирахме относителното изобилие от пътища в общите метагеноми 26, които бяха много по-постоянни и равномерно разнообразни, отколкото бяха организма (фиг. 2Б, виж също фиг. 1), потвърждавайки това като екологично свойство на целия човешки микробиом 3. По същия начин успяхме да установим за първи път, че таксономичното и функционалното алфа разнообразие в микробните общности значително корелират (Spearman на обратния r = 0,60 на Симпсън, p = 3,6e-67, n = 661), последният в по-забранен диапазон от конфигурации на общността (Supp. Фиг. 5).
Корелации с фенотип на гостоприемника
Наличието на пътеки и клади е най-съществено свързано (всички FDR q 1, въпреки че те все още трябва да включват внимателно съчетани вътрешни контроли. Уникалността на микробиома на всеки индивид дори в тази референтна популация аргументира бъдещите проучвания да обмислят перспективни вътрешно-обектни проекти, когато е възможно. Уникалната комбинация на HMP от организми и функционални данни за местообитанията на тялото, обхващаща както 16S, така и метагеномно профилиране, заедно с подробна характеристика на всеки субект, ни позволи и в следващите проучвания да преминем отвъд наблюдението на променливостта в човешкия микробиом, за да попитаме как и защо тези микробни общности се различават толкова широко.
Остават много подробности за по-нататъшна работа за попълване, надграждайки върху това референтно проучване. Как ранната колонизация и промяната през целия живот варират между местообитанията на тялото? Дали епидемиологичните модели на предаване на полезни или безвредни микроби отразяват моделите на предаване на патогени? Кои съпътстващи събития сред микробите отразяват споделен отговор на околната среда, за разлика от конкурентните или мутуалистични взаимодействия? Колко голяма роля играе имунитетът на домакините или генетиката при формирането на модели на разнообразие и как моделите, наблюдавани в това население на Северна Америка, се сравняват с тези по света? Бъдещите проучвания, базирани на каталозите на гените и организмите, създадени от Проекта за човешки микробиоми, включително все по-подробни изследвания на метатранскриптомите и метапротеомите, ще помогнат за разгадаването на тези отворени въпроси и ще ни позволят да разберем по-пълно връзките между човешкия микробиом, здравето и болестите.
Обобщение на методите
Взети са проби от микробиоми от до 18 места на тялото в един или два момента от 242 индивида, клинично изследвани за отсъствие на заболяване 2. Пробите бяха подложени на 16S rRNA генно пиросеквениране (454 Life Sciences) и подгрупа беше секвенирана за метагеномика с помощта на платформата Illumina GAIIx 1. Обработката на данни 16S и оценките на разнообразието бяха извършени с помощта на QIIME 27, а метагеномните данни бяха таксономично профилирани с помощта на MetaPhlAn 13, метаболитно профилирани от HUMAnN 26 и събрани за анотиране на гени и групиране в уникален каталог 1. Потенциалните патогени бяха идентифицирани с помощта на базата данни PATRIC 14, изолирани референтни анотации на генома, извлечени от KEGG 28, и референтно картографиране на генома, извършено от BWA 29, към намален набор от геноми, на които може да се съчетаят кратките четения 30. Микробните асоциации бяха оценени чрез мерки за сходство, отчитащи композицията 23, и тестове за фенотипна асоциация бяха проведени в R. Всички данни и допълнителни подробности за протокола са достъпни на http://hmpdacc.org. Пълните методи придружават тази статия.
Допълнителен материал
Благодарности
Консорциумът по човешки микробиом
Бележки под линия
1 Биостатистика, Харвардско училище за обществено здраве, Бостън, Масачузетс
2 Широкият институт на MIT и Харвард, Кеймбридж, Масачузетс
3 Катедра по химия и биохимия, Университет на Колорадо, Боулдър, Колорадо
4 Медицински институт на Хауърд Хюз, Боулдър, Колорадо
5 Институтът за геном, Медицинско училище във Вашингтонския университет, Сейнт Луис, Мисури
6 Институт J. Craig Venter, MD Rockville
7 Институт за геномни науки, Медицински факултет на Университета в Мериленд, д-р Балтимор
8 Център за секвениране на човешкия геном, Медицински колеж Baylor, Хюстън, Тексас
9 Катедра по патология и имунология, Медицински колеж Baylor, Хюстън, Тексас
10 Отделение по патология, Тексаска детска болница, Хюстън, Тексас
11 Катедра по акушерство и гинекология, Катедра по майчина фетална медицина, Медицински колеж Baylor, Хюстън, Тексас
12 Молекулярна и клетъчна биология, Университет на Гуелф, Гулеф, Канада
13 Катедра по гражданско и екологично инженерство, Масачузетски технологичен институт, Кеймбридж, Масачузетс
14 Център за биотехнологии в околната среда, Национална лаборатория Лорънс Бъркли, Бъркли, Калифорния
15 Стоматологично училище, Калифорнийски университет, Сан Франциско, Сан Франциско, Калифорния
16 Молекулярна вирусология и микробиология, Медицински колеж Baylor, Хюстън, Тексас
17 Национален институт по артрит и мускулно-скелетна и кожна болест, Национален институт по здравеопазване, д-р Бетесда
18 Служба за изследване на здравето на жените, Национални здравни институти, MD Bethesda
19 Национален институт по алергия и инфекциозни болести, Национален институт по здравеопазване, д-р Бетесда
20 Медицински департамент, Нюйоркски университет, Langone Medical Center, Ню Йорк, Ню Йорк
21 Национален институт за изследване на човешкия геном, Национален институт по здравеопазване, MD Bethesda
22 Департамент по статистически науки и оперативни изследвания, Университет на Вирджиния, САЩ, Ричмънд, Вирджиния
23 Център за изследване на биологичната сложност, Университет на Вирджиния в Британска общност, Ричмънд, Вирджиния
24 Катедра по биология, Университет на Британската общност, Ричмънд, Вирджиния
25 Група за технологична интеграция, Национален център за научни изчисления в областта на енергийните изследвания, Национална лаборатория Лорънс Бъркли, Бъркли, Калифорния
26 Genome Science Group, Bioscience Division, Los Alamos National Laboratory, Los Alamos NM
27 Joint Genome Institute, Walnut Creek CA
28 Център за управление на биологични данни и технологии, Отдел за изчислителни изследвания, Национална лаборатория Лорънс Бъркли, Бъркли, Калифорния
29 Национален институт по дентални и черепно-лицеви изследвания (NIDCR), Национален институт по здравеопазване, д-р Бетесда
30 FemCare Продуктови въпроси за безопасност и регулиране, The Procter & Gamble Company, Cincinnati OH
31 Биоинформатичен отдел, Second Genome, Inc., Сан Бруно, Калифорния
32 Департамент по молекулярна генетика, Институт Форсайт, Кеймбридж, Масачузетс
33 Катедра по орална медицина, инфекции и имунитет, Харвардско училище по дентална медицина, Бостън, Масачузетс
34 Катедра по медицина, Отдел по обща медицинска наука, Медицинско училище във Вашингтон, Сейнт Луис, Мисури
35 Катедра по патология и имунология, Медицински факултет на Вашингтонския университет, Сейнт Луис, Мисури
36 bioMerieux, Inc., Durham NC
37 drive5.com, Тибурон, Калифорния
38 Център за етика, хуманитарни и духовни грижи, клиника в Кливланд, Кливланд, Охайо
39 Катедра по структурна биология, VIB, Белгия, Брюксел, Белгия
40 Департамент по приложни биологични науки (DBIT), Vrije Universiteit Brussel, Брюксел, Белгия
41 Катедра по биоинформатика и геномика, Университет на Северна Каролина - Шарлот, Шарлот, Северна Каролина
42 Катедра по биологични науки, Университет в Айдахо, Москва ID
43 Изчислителна и системна биология, Масачузетски технологичен институт, Кеймбридж, Масачузетс
44 Център за напреднало дентално образование, Университет Сейнт Луис, Сейнт Луис МО
45 Катедра по компютърни науки, Университет в Колорадо, Боулдър, Колорадо
46 Отдел за асоциирани клинични специалитети и Институт по дентални изследвания, UCLA School of Dentistry, Лос Анджелис, Калифорния
47 University of Maryland Francis King Carey School of Law, д-р Балтимор
48 Център Paul Josephine Bay, Морска биологична лаборатория, Уудс Хоул, Масачузетс
49 Отдел по екология, Отдел за науките за Земята, Национална лаборатория Лорънс Бъркли, Бъркли, Калифорния
50 Катедра по пародонтология, Университет в Тексас, Център за здравни науки, Стоматологично училище, Хюстън, Тексас
51 Катедра по биология, Държавен университет в Сан Диего, Сан Диего, Калифорния
52 Медицински факултет, Университет Макгил, Монреал, Канада
53 Дерматологичен клон, CCR, Национален институт по рака, Bethesda MD
54 Катедра по микробиология, Университет Корнел, Итака, Ню Йорк
55 Център за биоинформатика и изчислителна биология, Университет в Мериленд, MD College College
56 Отдел по инфекциозни болести, Детска болница в Бостън, Медицинско училище в Харвард, Бостън, Масачузетс
57 Департамент по антропология, Университет в Оклахома, Норман ОК
58 Катедра по акушерство и гинекология, Медицинско училище във Вашингтонския университет, Сейнт Луис, Мисури
59 Отдел по гастроентерология и хепатология, Университет на Алабама в Бирмингам, Бирмингам, Алабама
60 Център за медицинска етика и здравна политика, Медицински колеж Baylor, Хюстън, Тексас
61 Медицинско-инфекциозна болест, Медицински колеж Baylor, Хюстън, Тексас
62 Biosciences Division, Oak Ridge National Laboratory, Oak Ridge TN
63 Институти Гладстоун, Калифорнийски университет, Сан Франциско, Сан Франциско, Калифорния
64 Институт за хуманна генетика, Калифорнийски университет, Сан Франциско, Сан Франциско, Калифорния
65 Отдел по биостатистика, Калифорнийски университет, Сан Франциско, Сан Франциско, Калифорния
66 Катедра по компютърни науки, Университет в Мериленд, MD College College
67 Училище по информатика и компютърни науки, Университет Индиана, Блумингтън IN
68 Медицинско училище Mount Mount Sinai, Ню Йорк, Ню Йорк
69 Молекулярна и човешка генетика, Медицински колеж Baylor, Хюстън, Тексас
70 Център за биоетика и Катедра по медицинска етика, Университет на Пенсилвания, Филаделфия, Пенсилвания
71 Катедра по микробиология и имунология, Университет в Мичиган, Ан Арбър, Мичиган
72 Катедра по микробиология и молекулярна генетика, Мичигански държавен университет, Източен Лансинг, Мичиган
73 EMMES Corporation, MD Rockville
74 Университетска болница Харпър, Медицински факултет на държавния университет Уейн, Детройт, Мичиган, Детройт, Мичиган
75 Институт по генетична медицина McKusick-Nathans, Медицински факултет на Университета Джон Хопкинс, д-р Балтимор
76 Институт J. Craig Venter, Сан Диего, Калифорния
77 Медицинско училище Файнберг, Северозападен университет, Чикаго, IL
78 Alkek Center for Metagenomics and Microbiome Research, Baylor College of Medicine, Houston TX
Допълнителната информация е свързана с онлайн версията на статията на адрес www.nature.com/nature.
Принос на автора
Основни изследователи: BWB, RAG, SKH, BAM, KEN, JFP, GMW, OW, RKW Подготовка на ръкописа: DG, CH, RK, OW
Управление на финансиращата агенция: CCB, TB, VB, JLC, SC, CD, VDF, CG, MYG, RDL, JM, PM, JP, LMP, JAS, LW, CW, KAW
Ръководство на проекта: SA, JHB, BWB, ATC, HHC, AME, MGF, RSF, DG, MGG, KH, SKH, CH, EAL, RM, VM, JCM, BAM, MM, DMM, KEN, JFP, EJS, JV, GMW, OW, AMW, KCW, JRW, SKY, QZ
Подготовка за анализ на ръкопис: JCC, KF, DG, AG, KHH, CH, RK, DK, HHK, OK, KPL, REL, JR, JFS, PDS, NS
Освобождаване на данни: LA, TB, IAC, KC, HHC, NJD, DJD, AME, VMF, LF, JMG, SG, SKH, MEH, CJ, VJ, CK, AAM, VMM, TM, MM, DMM, JO, KP, JFP, CP, XQ, RKS, NS, IS, EJS, DVW, OW, KW, KCW, CY, BPY, QZ
Методи и изследователска разработка: SA, HMA, MB, DMC, AME, RLE, MF, SF, MGF, DCF, DG, GG, BJH, SKH, MEH, WAK, NL, KL, VM, ERM, BAM, MM, DMM, CN, JFP, MEP, XQ, MCR, CR, EJS, SMS, DGT, DVW, GMW, YW, KMW, SY, BPY, SKY, QZ
Производство на ДНК последователност: SA, EA, TA, TB, CJB, DAB, KDD, SPD, AME, RLE, CNF, SF, CCF, LLF, RSF, BH, SKH, MEH, VJ, CLK, SLL, NL, LL, DMM, IN, CN, MO, JFP, XQ, JGR, YR, MCR, DVW, YW, BPY, YZ
Събиране на клинични проби: KMA, MAC, WMD, LLF, NG, HAH, ELH, JAK, WAK, TM, ALM, PM, SMP, JFP, GAS, JV, MAW, GMW
Експерти на сайта на тялото: KMA, EAV, GA, LB, MJB, CCD, FED, LF, JI, JAK, SKH, HHK, KPL, PJM, JR, TMS, JAS, JDS, JV
Етични, правни и социални последици: RMF, DEH, WAK, NBK, CML, ALM, RR, PS, RRS, PS, LZ
Управление на напрежението: EAV, JHB, IAC, KC, SWC, HHC, TZD, ASD, AME, MGF, MGG, SKH, VJ, NCK, SLL, LL, KL, EAL, VMM, BAM, DMM, KEN, IN, IP, LS, EJS, CMT, MT, DVW, GMW, AMW, YW, KMW, BPY, LZ, YZ
16S анализ на данни: KMA, EJA, GLA, CAA, MB, BWB, JPB, GAB, SRC, SC, JC, TZD, FED, ED, AME, RCE, MF, AAF, JF, HG, DG, BJH, TAH, SMH, CH, JI, JKJ, STK, SKH, RK, HHK, OK, PSLR, REL, KL, CAL, DM, BAM, KAM, MM, MP, JFP, MP, KSP, XQ, KPR, MCR, BR, PDS, TMS, NS, JAS, WDS, TJS, CSS, EJS, RMT, JV, TAV, ZW, DVW, GMW, JRW, KMW, YY, SY, YZ
Обработка и подравняване на данни от пушка: CJB, JCC, ED, DG, AG, MEH, HJ, DK, KCK, CLK, YL, JCM, BAM, MM, DMM, JO, JFP, XQ, JGR, RKS, NUS, IS, EJS, GGS, SMS, JW, ZW, GMW, OW, KCW, TW, SKY, LZ
Асамблея: HMA, CJB, PSC, LC, YD, SPD, MGF, MEH, HJ, SK, BL, YL, CL, JCM, JMM, JRM, PJM, MM, JFP, MP, MEP, XQ, MR, RKS, MS, DDS, GGS, SMS, CMT, TJT, WW, GMW, KCW, LY, YY, SKY, LZ
Анотация: OOA, VB, CJB, IAC, ATC, KC, HHC, ASD, MGG, JMG, JG, AG, SG, BJH, KH, SKH, CH, HJ, NCK, RM, VMM, KM, TM, MM, JO, KP, MP, XQ, NS, EJS, GGS, SMS, MT, GMW, KCW, JRW, CY, SKY, QZ, LZ
WGS метаболитна реконструкция: SA, BLC, JG, CH, JI, BAM, MM, BR, AMS, NS, MT, GMW, SY, QZ, JDZ
Информация за автора
Всички данни, използвани в това проучване, са достъпни от Центъра за анализ и координация на данни за човешки микробиом 1. Информацията за препечатки и разрешения е достъпна на www.nature.com/reprints.
Авторите не декларират конкуриращи се финансови интереси.
- Затлъстяването променя мозъчната структура и функция Science The Guardian
- Растителни храни и средиземноморска диета, свързани със здравословен чревен микробиом EurekAlert! Наука
- Определение на стомаха, функция, структура, диаграма и факти Британика
- Захар, мазнини, протеини; Нишестета, използвани в SF Gate за здравословно хранене на човешкото тяло
- Метаболитно здравословно затлъстяване и риск от намаляване на бъбречната функция - Чанг - 2018 - Затлъстяване - Wiley