Алехандро Манзано-Марин
UMR 1062 Center of Biologie pour la Gestion des Populations, INRA, CIRAD, IRD, Montpellier SupAgro, Univ. Монпелие, 755 avenue du campus Agropolis, Монпелие, Франция
Свързани данни
Резюме
Задължителните симбиотични асоциации присъстват в голямо разнообразие от животни с диета с ограничени хранителни вещества. Листни въшки (hemiptera: Aphididae) почти универсално приемат бактерии Buchnera aphidicola в специализирани органи (наречени бактериоми). Тези бактерии снабдяват листната въшка с основни хранителни вещества, които липсват в диетата им (т.е. основни аминокиселини и някои витамини от група В). Някои линии на листни въшки, като видове от подсемейство Lacninae, са се развили като съвместни асоциации с вторични ендосимбионти, произтичащи от загубата на биотин и рибофлавин-биосинтетични гени. В това проучване анализирам повторно публикувани преди това данни за секвениране от бананова листна въшка Pentalonia nigronervosa. Доказвам, че метаболитното заключение е резултат от De Clerck et al. (Microbiome 3:63, 2015) са неправилни и е възможно да възникнат от използването на неадекватни методи. Освен това обсъждам как привидно предубедената интерпретация на техните анализи за лечение на антибиотици, заедно с неправилен метаболитен извод, основан на генома, доведе до погрешното предположение „че в банановата листна въшка възниква съзадължителна симбиоза между B. aphidicola и Wolbachia“.
Основен текст
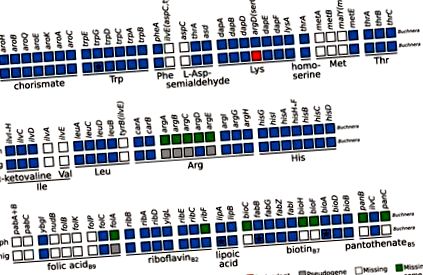
Основни аминокиселини и избрани витамин В и биосинтетични метаболитни способности на витамин В на задължителни симбиотични консорциуми от различни видове листни въшки Диаграма, обобщаваща метаболитните възможности на Buchnera от „моносимбиотични“ Aphidinae листни въшки (Aph) и P. nigronervosa (Pnig). Редовете „Aph“ са свито представяне на няколко вида Aphidinae (виж Допълнителна таблица S3 в Допълнителен файл 1). Като имена на колони се използват имената на гени, кодиращи ензими, участващи в биосинтетичния път. Кутиите на всеки ред представляват гените, кодирани от генома на Buchnera. В долната част, линии, подчертаващи гените, участващи в пътя, водещ до съединението, посочено от името под линията. За аминокиселините се използва неговото трибуквено съкращение
Заключение
Накратко, De Clerck et al. [1] не предоставят доказателства, че очевидното фиксиране на Wolbachia в анализираните популации на P. nigronervosa се дължи на това, че този симбионт е хранителен съ-облигат в тази листна въшка. Повторният анализ на данните за секвениране разкрива, че заключенията, основани на генома, за хранителна съ-облигатна симбиоза при P. nigronervosa са били погрешни. Освен това проверката на праймерните последователности разкри, че повечето двойки праймери не съвпадат с целевите последователности в Buchnera от P. nigronervosa. И накрая, резултатите от антибиотичното лечение имат алтернативни обяснения, които авторите не са успели да обсъдят и които не могат да бъдат отхвърлени без по-чувствителни анализи. Независимо от това, очевидно постоянното присъствие на Wolbachia все пак може да бъде обяснено с други процеси, като например някаква репродуктивна манипулация, защита срещу вирусни инфекции или извадки от листни въшки, които действително принадлежат към един или няколко „суперклона (и)“, първоначално заразени с тясно свързани ендосимбионти на Wolbachia.
Материали и методи
Последователно събиране на данни и сглобяване на генома
Библиотеките за секвениране на дървета (SRR1661114, SRR1662246, SRR1662249) от De Clerck et al. [1] са изтеглени от базата данни на NCBI SRA (присъединяване SRX766492) от BioProject (https://www.ncbi.nlm.nih.gov/bioproject/268300, последен достъп на 15 април 2019 г.). Проверката на изтеглените файлове fastq с fastQC v0.11.7 (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/, последно достъпно на 15 април 2019 г.) разкри замърсяване на четенията с индекси на адаптера TruSeq 4, 5 и 6, както и неизвестен T-G-богат адаптер („TGTGTTGGGTGTGTTGGGTGTGTTGGGTGTGTTGGGTGTGTTGGGTGTGT“). За да отрежа тези адаптери от прочетените набори от данни и да извърша качествено подрязване, използвах fastx_clipper, последван от fastx_quality_trimmer, който извърши качествено подрязване вдясно (използвайки минимален качествен праг от 20) и четене по-малко от 50 bp бяха отпаднали (опции -t 20 -l 50). Освен това PRINSEQ v0.20.4 [10] беше използван за премахване на четения, съдържащи недефинирани нуклеотиди, както и за отделяне на останалите без двойка след процеса на филтриране и подрязване. Сглобяването на трите библиотеки беше направено с помощта на SPAdes v3.10.1 [11] с k-mers 33, 55 и 77.
Събиране на сглобени последователности към Бухнера и Волбахия
Първо, всички скелета, по-къси от 200 bp и със средно k-mer покритие по-ниско от 3, бяха отпаднали. Това доведе до 10 097 (3,74% от общия брой скелета), които след това бяха използвани за BLASTX-базирани биннинг. Това беше направено чрез изграждане на база данни за протеомите на граховата листна въшка и набор от бактерии Wolbachia и Buchnera. Изградена е отделна база данни за митохондрионните протеини, кодирани от листната въшка Schizaphis graminun, за да се открият митохондрийните последователности. Всички тези набори от данни за протеини са били достъпни преди времето на публикуване на De Clerck et al. [1]. Номерата за присъединяване и информация за щамовете могат да бъдат намерени в допълнителна таблица S4 (Допълнителен син файл 1). Търсенето на BLASTX срещу гореспоменатите бази данни беше извършено с помощта на филтрираните скелета като заявка (-soft_masking true -seg yes -max_target_seqs 10000 -evalue 1e-3), последвано от присвояване на скелето на организма с най-добрия BLASTX хит на всяко скеле. След това тези кошчета бяха филтрирани, като се зададе минимален праг на покритие от k-mer от 10 както за Buchnera, така и за Wolbachia. Това доведе до 135 и 1309 скелета, съответно на всяка бактерия.
Идентифициране на „липсващи“ и „хранителни гени“ в Buchnera
Пригодността на докладваните праймери в De Clerck et al. [1] (намерен в „Допълнителен файл 1: Таблица S1“) се оценява чрез подравняването им спрямо идентифицираната и докладвана генна последователност в настоящата работа, използвайки AliView v1.26 [13].
- Нови подходи към хранителната терапия
- Хранителни факти за картофи Здравословно хранене SF Gate
- Ново доказателство, че терапията с тестостерон е ефективно лечение на затлъстяването при мъжете
- Политика за етикетиране на менюто на ресторанта Преглед на доказателства и противоречия
- Болест на малките кораби Джоана Уордлоу разглежда доказателствата Възраст Великобритания