Пола В. Уеландър
департамент по биология, Масачузетски технологичен институт, 77 Massachusetts Avenue, 68-380, Cambridge, MA 02139;
Морийн Л. Колман
департамент по биология, Масачузетски технологичен институт, 77 Massachusetts Avenue, 68-380, Cambridge, MA 02139;
Алекс Л. Сесии
b Отдел по геоложки и планетарни науки, Калифорнийски технологичен институт, Пасадена, MC100-23, 1200 East California Boulevard, Pasadena, CA 91125;
Роджър Е. Призоваване
c Департамент по земни, атмосферни и планетарни науки, Масачузетски технологичен институт, 77 Massachusetts Avenue, E25-633, Кеймбридж, Масачузетс 02139; и
Даян К. Нюман
департамент по биология, Масачузетски технологичен институт, 77 Massachusetts Avenue, 68-380, Cambridge, MA 02139;
c Департамент по земни, атмосферни и планетарни науки, Масачузетски технологичен институт, 77 Massachusetts Avenue, E25-633, Кеймбридж, Масачузетс 02139; и
d Медицински институт Howard Hughes, 77 Massachusetts Avenue, 68-171, Cambridge, MA 02139
Свързани данни
Резюме
Резултати
За да определим кой ген е необходим за метилиране на С-2, първо идентифицирахме предполагаеми биосинтетични гени на хопаноиди в генома на R. palustris TIE-1. Понастоящем единственият окончателно известен хопаноиден биосинтетичен етап е първоначалната циклизация на сквален до основната структура на хопена диплоптен от сквален-ходен циклазата (shc). Предишно проучване в Zymomonas mobilis и Bradyrhizobium japonicum идентифицира няколко предполагаеми гена за биосинтеза на хопаноиди, заобикалящи локуса на shc (24). Изследвахме регионите нагоре и надолу по веригата на shc локуса в TIE-1 и открихме подобен предполагаем хопаноиден биосинтетичен генен клъстер (Фигура 1 А). Приблизително девет килобази (kb) нагоре от гена на сквален-хоуден циклазата наблюдавахме отворена рамка за четене (ORF 4269), която беше анотирана като предполагаем B-12 свързващ радикален SAM протеин (Фиг. 1 А). Тъй като е показано, че клетките на R. palustris произвеждат 2-метилхопаноиди, маркирани в позицията С-2, когато се хранят с етикет метионин (20, 25), изглежда разумно това метилиране да се осъществи чрез механизъм, зависим от S-аденозилметионин (SAM). По този начин присъствието на радикален SAM мотив в ORF 4269 го прави привлекателен кандидат за метилазата.
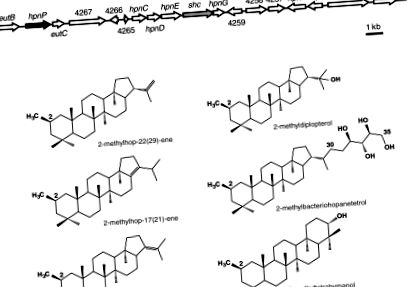
Идентифициране на предполагаем хостеоиден биосинтетичен генен клъстер в R. palustris TIE-1. (А) Локусът shc (Сива стрелка), който е необходим за биосинтеза на хопаноиди, е заобиколен от няколко предполагаеми гена за биосинтез на хопаноиди. Преди shc е идентифициран хипотетичен B-12 свързващ радикален SAM протеин (hpnP, Black Arrow) като кандидат за C-2 метилаза. (Б) Структурата на шестте С-2 метилирани тритерпеноиди, произведени от R. palustris TIE-1: три 2-метилхопени (хмел-22 (29) -ен, хмел-17 (21) -ен и хмел-21- ен), 2-метилдиплоптерол, 2-метилтетрахиманол и 2-метилбактериохопантетрол.
ORF 4269 беше изтрит в рамка чрез използване на хомоложна рекомбинационно медиирана генна делеция и общите липидни екстракти бяха анализирани чрез газова хроматография-масова спектрометрия (GC-MS) за производството на метилирани хопаноиди. Доказано е, че R. palustris TIE-1 произвежда поне шест различни С-2 метилирани тритерпеноиди (фиг. 1 В) и полученият щам на делеция не произвежда нито една от тези молекули (фиг. 2 и фиг. S1). Производството на С-2 метилирани хопаноиди се възстановява чрез предоставяне на копие на гена на R. palustris метилаза под естествения промотор върху самовъзпроизвеждащ се плазмид (Фиг. 2). Следователно ORF 4269 е единственият ген, необходим за метилиране в позиция C-2 и предлагаме локусът да бъде преименуван на hpnP, следвайки номенклатурата, установена в Z. mobilis и B. japonicum (24).
GC-MS общо йонни хроматограми на ацетилирани екстракти от общи липиди от култури R. palustris. Мутантът ΔhpnP не произвежда C-2 метилирани хопаноиди. (A) R. palustris TIE-1. (B) R. палустрис ΔhpnP. (C) R. palustris ΔhpnP, допълнен с гена hpnP върху самовъзпроизвеждащ се плазмид. Номерирани съединения: I, 2-метилхопени; II, надежда; III, 2-метилдиплоптерол; IV, диплоптерол; V, 2-метилтетрахиманол; VI, тетрахиманол; VII, 2-метилбактериохопанететрол; и VIII, бактериохопантетрол. Ацетилирани общи липидни екстракти бяха анализирани чрез високотемпературна GC-MS и съединенията бяха идентифицирани чрез сравняване на времената на задържане и масспектрите с автентични съединения и публикувани мас спектри (Таблица S6). Peak II е съвместно елуиране на две надеждни структури, hop-22 (29) -ene и hop-21-ene. Не се елуират метилирани хопаноиди между 29–42 минути (пълна хроматограма, показана на фиг. S1). Комплементираният ΔhpnP щам наистина произвежда 2-метилбактериохопанететрол (съединение VII), както е потвърдено чрез анализ на 205 Da масова хроматограма и мас спектъра; пикът обаче е твърде широк, за да се види при разделителната способност на тази цифра на общия йонния ток на GC-MS.
Поява на гени на сквален-хоуден циклаза (shc) и hpnP метилаза в далечно свързани бактерии. Дървото представлява филогенетични взаимоотношения между бактериалните видове и е конструирано с максимална вероятност, използвайки пет конкатенирани основни протеина (Adk, RpoB, GyrB, RecA и SecY). Сините ленти във вътрешния пръстен показват щамове с shc; Червените ленти във външния пръстен показват щамове с hpnP. Изобразени са всички секвенирани изолати, съдържащи shc и/или hpnP, както и представители от други бактериални семейства. Родовете Bacillus (34 изолата) и Burkholderia (58 изолата) са сринати.
Както може да се види в Таблица S1, организмите, които съдържат метилазата, се намират в различни среди, включително сладка вода, почва, отпадъчни води, стъблени възли и коренови възли. В съответствие с рядкостта на 2-метилхопаноидите в култивирани морски цианобактерии (17), нито една от секвенираните цианобактерии, които съдържат гена на метилазата, не е изолирана от морска среда. Тъй като се смята, че по-голямата част от седиментните 2-метилхопани произхождат от древни плитки морски среди, доминирани от фототрофи (18, 19), ние очаквахме, че нашият биоинформатичен анализ може да разкрие наличието на HpnP метилаза в все още непроверена морска цианобактерия. Въпреки това, α-протеобактерията Nitrobacter sp. Nb-311A е единственият морски организъм както в геномната, така и в морската метагеномна база данни, съдържащ хомолог на HpnP метилазата.
За да разберем произхода на този протеин, реконструирахме филогенията му с максимална вероятност и тествахме чувствителността на топологията към изравняването на входа и избора на таксони извън групата. Моделът за заместване на LG (29), избран от ProtTest (30), беше използван за всички опити и бяха използвани 10 произволни стартови дървета, за да се предотврати търсенето на дървета от задържане в локален оптимум. Тъй като качеството на подравняването е основен фактор, определящ точността на дървото (31, 32), използвахме три програми за подравняване (CLUSTALW, Muscle и T-Coffee) (33–35). Освен това използвахме три стратегии за филтриране (спокойна, строга и без филтриране), за да премахнем несигурни позиции за подравняване (фиг. S2). Въпреки че филтрирането може да увеличи съотношението сигнал/шум на данните, то също така изхвърля потенциално информативни сайтове и може да не е от полза за кратки изравнения (36, 37). За да вкореним дървото, използвахме всички членове на сестра клада от радикални SAM протеини като извънгрупа (Фиг. S3), подход, предложен като оптимален (38). За сравнение добавихме втори клад извън група в някои опити (фиг. S3).
Некоренованата топология на групата показва различни клъстери за протеина HpnP от видовете Methylobacterium, групата Bradyrhizobium/Nitrobacter/Rhodopseudomonas и цианобактериите (фиг. S4). Редът на разклоняване на тези групи обаче варира значително между опитите. Появиха се три различни вкоренени топологии (фиг. 4 и таблица S3). Най-често α-протеобактериалните HpnP последователности са монофилетични и са вложени в редици от цианобактериални HpnP последователности и от тази топология е най-скромен да се направи изводът, че родовия HpnP е пребивавал в цианобактерия (фиг. 4 А). В някои случаи цианобактериалните и а-протеобактериалните последователности образуват сестринни платна (фиг. 4 В) или цианобактериалните последователности са вложени в а-протеобактериалните последователности (фиг. 4 С). Тази неяснота предполага, че има недостатъчен филогенетичен сигнал, който да разреши уверено реда на разклоняване на основните кладове. Тествахме дали най-добрата топология, възстановена за дадено подравняване, е значително по-добра от двете конкуриращи се топологии, използвайки теста Приблизително безпристрастен (39). Този тест не успя да отхвърли нито една от конкуриращите се хипотези (Таблица S4) и по този начин дори за дадено подравняване не можем уверено да изберем най-доброто дърво.
Филогенетични връзки между HpnP метилази от три отдалечено свързани бактериални фили. (А) Най-доброто дърво, намерено в опит 15 (Таблица S3), с ранно разклоняващо се цианобактериално HpnP. (B) Най-доброто дърво в девет опит, с монофилетичен цианобактериален HpnP. (C) Най-доброто дърво в опит две, с ранно разклоняване Methylobacterium HpnP. Поддръжката на клон се показва от непараметрични статистически данни за приблизително съотношение на вероятност (aLRT). Съкращения на рода: M = Methylobacterium, N = Nitrobacter, B = Bradyrhizobium и R = Rhodopseudomonas.
Независимо от точния ред на разклоняване, нашите резултати разкриват сложна еволюционна история за HpnP. Неговото филетично разпределение може да възникне по два начина: или е присъствал в прародителя на цианобактериите и α-протеобактериите и е бил многократно изгубен, или е бил хоризонтално прехвърлен между цианобактерии и α-протеобактерии. Наблюдавано е прехвърляне на гени между тези фили (40) и цианобактериите най-често споделят гени с а-протеобактериите в сравнение с други фили (41). В рамките на цианобактериите HpnP се разпространява спорадично и филогенията HpnP не съответства на филогенията на вида (42). Това предполага трансфер на гени и загуба на ген сред цианобактериите и по този начин е невъзможно да се обобщи дали всички древни цианобактерии са произвеждали 2-метилхопаноиди. Позицията на самотната ацидобактерия в HpnP дървото също е несигурна. В рамките на α-протеобактериите HpnP и видовите филогении са сходни (43), което предполага вертикално спускане в рамките на тази група. От този филогенетичен анализ става ясно, че намирането на повече таксони за попълване на HpnP дървото е от решаващо значение за по-доброто разрешаване на историята на този протеин.
Дискусия
В това проучване идентифицирахме радикална SAM метилаза, кодирана от hpnP, която е необходима за C-2 метилиране на бактериални хопаноиди. Понастоящем генът hpnP изглежда силен предиктор за способността да произвежда 2-метилхопаноиди; дали ще остане изключителен предиктор, предстои да разберем, тъй като в бъдеще ще бъдат идентифицирани повече гени на метилаза. Засега обаче използването на 2-метилхопаноиди и утаечни 2-метилхопани като индикатори за цианобактерии ни подтикна да направим оценка на разпределението на геновия продукт hpnP в бактериални геноми. Установихме, че присъствието на HpnP не е ограничено до цианобактериите, тъй като е открито и в α-протеобактериите (по-специално rhizobiales) и едната ацидобактерия.
В заключение, четири реда доказателства предполагат, че 2-метилхопаните не могат да се използват като биомаркери за кислородна фотосинтеза: i) hpnP се разпределя в много съвременни бактерии, които не участват в кислородната фотосинтеза, ii) цианобактериите не са задължителни кислородни фотоавтотрофи (51), iii) не всички цианобактерии правят 2-метилхопаноиди (17) и iv) в цианобактерията Nostoc punctiforme, 2-метилхопаноидите се локализират във външните мембрани на акинети, структура за оцеляване, която не е фотосинтетично активна (23). Следователно разрешаването на биологичната функция на 2-метилхопаноидите в съвременните бактерии е необходимо, за да се интерпретира по-добре значението на 2-метилхопаните в древните седименти. Независимо от тяхната клетъчна функция, прилагането на (фило) генетика за идентифициране и интерпретиране на историята на гена, необходим за биосинтеза на конкретно геостабилно съединение, показва как два класа молекулни вкаменелости - гени и биомаркери - могат да се ограничат и да осигурят по-информирано разбиране за микробната еволюция и метаболизма.
Материали и методи
Бактериални щамове и условия за растеж.
Бактериалните щамове, използвани в това проучване, са изброени в таблица S5. Щамовете на Escherichia coli се отглеждат в лизогенен бульон (LB) при 37 ° С. Щамовете Rhodopseudomonas palustris се отглеждат хемохетеротрофно в небуферирана YP среда (0,3% екстракт от дрожди, 0,3% пептон) при 30 ° С на тъмно, докато се разклаща при 250 RPM. За растеж върху твърда среда LB или YP се втвърдяват с 1.5% агар и се допълват, ако е необходимо, с гентамицин при 20 μg/mL (E. coli) или 800 μg/mL (R. palustris).
ДНК методи, плазмидна конструкция, трансформация и деформация.
Всички плазмидни конструкции са описани в таблица S5. QIAprep Spin Miniprep Kit (Qiagen) се използва за изолиране на плазмидна ДНК от Е. coli. Геномна ДНК от щамове R. palustris се изолира с помощта на DNeasy Blood and Tissue Kit (Qiagen). ДНК последователностите на всички междинни продукти за клониране са потвърдени чрез секвениране в лабораторията за биополимери в Центъра за изследване на рака в MIT. Щамовете на Е. coli се трансформират чрез електропорация чрез използване на Electroporator 2510 (Eppendorf), както се препоръчва от доставчика. Плазмидите бяха мобилизирани от E. coli S17-1 в R. palustris чрез конюгиране върху YP агарови плаки, които бяха инкубирани една нощ при 30 ° С (52, 53). Делецията на локуса на hpnP в TIE-1 и допълването на мутанта на делеция се извършват, както е описано по-рано (15).
Анализ на газова хроматография-масспектрометрия (GC-MS).
За хопаноиден анализ щамовете бяха отгледани до късна стационарна фаза (7 d). Общите липидни екстракти се приготвят чрез екстракция с разтворител, дериватизират се като ацетати и се анализират чрез високотемпературна GC-MS, като се използва DB-XLB колона, както е описано по-горе (15). Съединенията бяха идентифицирани чрез сравняване на времената на задържане и масспектрите с автентични съединения (тетрахиманол от Trimyema sp .; диплоптерол от Methylococcus capsulatus; бактериохопанпентол от M. capsulatus) и публикувани мас спектри [hopenes: (54); тетрахиманол: (55)]. С-2 метилирането беше изведено от изместването на фрагмента m/z 191 до 205, m/z 369 до 383 и от относителното време на задържане в сравнение с дезметиловия хомолог (~ 0,15 минути по-рано). Времената на задържане и молекулните йони са изброени в таблица S6.
Филогения на HpnP.
Хомолозите на HpnP бяха идентифицирани чрез BLAST и подравнени с помощта на ClustalW, Muscle или T-Coffee (33–35). Подравняванията бяха изрязани от Gblocks (56), използвайки строги (параметри по подразбиране) или спокойни настройки. За строгия случай не се допускаха пропуски във всяка последователност. За спокойния случай минималният брой последователности за запазени и флангови позиции бяха зададени на минималните им допустими стойности, максималният брой на неконсервираните позиции беше зададен на 100, минималната дължина на блок беше 4 и бяха позволени пропуски в половината последователности. Фигура S2 показва примерно подравняване, генерирано от Muscle и изрязано от Gblocks. Уместността на двете семейства извън групата беше потвърдена от по-изчерпателно дърво от свързани протеини (Фиг. S3), което ясно показва, че HpnP и семействата извън групата са тясно свързани, но различни кладове.
Дърветата с максимална вероятност са конструирани от PhyML (29), използвайки LG + гама модел [избран от ProtTest (30)], 6 категории гама скорост, 10 произволни стартови дървета, размяна на SPR + NNI клонове и параметри на заместване, изчислени от данните. Поддръжката на клонове се определя от приблизителна статистика на LRT, подобна на SH (29). Видовото дърво е конструирано по подобен начин, като се използват пет конкатенирани протеинови последователности (RpoB, RecA, Adk, GyrB и SecY) от 375 вида, изрязани с помощта на Gblocks до 1679 позиции и вкоренени в Firmicutes. Видовото дърво не е предназначено да бъде изчерпателно, но се използва за показване на разпределението на гените shc и hpnP в бактериалната фила.
Използвахме Приблизително безпристрастен тест (39), реализиран в CONSEL (57), за да сравним топологиите на дърветата. Топологиите, показани на фиг. 4, бяха използвани като ограничения, а дължините на разклоненията и параметрите на заместване бяха оптимизирани върху тези топологии от PhyML. След това се сравнява вероятността за най-доброто необвързано дърво с вероятностите за двете алтернативни дървета, като се дава р-стойност на AU тест за всяко дърво (Таблица S4). Всяко дърво с р-стойност по-малка от 0,05 може да бъде отхвърлено. Ако няколко дървета имат p-стойности над тази граница, тогава не можем да изберем вярно истинското дърво измежду тях.
Допълнителен материал
Благодарности.
Благодарим на Дейвид Даути, Джейкъб Валдбауер и Ерик Алм за техническа помощ и полезни дискусии, както и на рецензентите за техните конструктивни коментари, които подобриха ръкописа. Тази работа беше подкрепена от безвъзмездни средства от Националната програма за аеронавтика и космическо управление (НАСА) за екзобиология (ALS, DKN и RES), Института по астробиология на НАСА (RES), Национална научна фондация Постдокторска стипендия за малцинства (PVW) и Институт Агурон Постдокторска стипендия по геобиология (MLC). Д.К.Н. е изследовател от Медицинския институт Хауърд Хюз.
Бележки под линия
Авторите не декларират конфликт на интереси.
- Идентифициране на 130 kDa клетъчна повърхност LDL-свързващ протеин от гладкомускулни клетки като частичен
- Как да конфигурирам консул в производствена среда на Ubuntu DigitalOcean
- IJMS Безплатен пълнотекстов Aedes albopictus, свързан с автофагия, ген 8 (AaAtg8) е необходим, за да се свърже
- Производство на метаболитна топлина - общ преглед на ScienceDirect теми
- Недостатъчният прием на никотинова киселина може да има сериозни последици за здравето