Резюме
Заден план
Метагеномните изследвания на човешката микробиота стават все по-широко разпространени в академичните изследвания, както и в хранителната и фармацевтичната индустрия и клиничния контекст. Интуитивните инструменти за изследване на експериментални данни представляват голям интерес за изследователите.
Резултати
Knomics-Biota е уеб базиран ресурс за изследователски анализ на метагеномите на човешките черва. Потребителите могат да генерират и споделят аналитични отчети, съответстващи на често срещани експериментални схеми (като изследване на случай-контрол или сдвоено сравнение). Интерактивните визуализации и статистически анализ се предоставят във връзка с външните фактори и в контекста на хиляди публично достъпни набори от данни, подредени в тематични колекции. Уеб услугата е достъпна на адрес https://biota.knomics.ru.
Заключения
Уеб услугата Knomics-Biota е изчерпателен инструмент за интерактивен анализ на метагеномни данни.
Заден план
Изпълнение
Изчислителната бекенда на системата се намира в облака (Допълнителен файл 1: Фигура S1) и използва публично достъпни софтуерни решения. Интерфейсът на интерфейса на уеб услугата е реализиран с помощта на Yii framework, а интерактивните визуализации се основават на d3js библиотека. Уеб услугата се предлага на адрес: https://biota.knomics.ru. След като се регистрира, потребителят може да качи свои собствени метагеномни набори за четене (получени с помощта на 16S rRNA или „пушка“/WGS [целият геном] секвениране), придружени с файлове за описание на данни (метаданни).
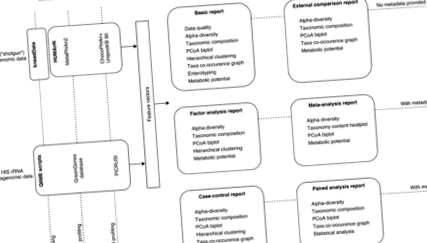
Общата логика на услугата Knomics-Biota включва два компонента: първичен и вторичен анализ (фиг. 1). Първичният компонент за анализ обхваща основна обработка на показанията за получаване на профили на състава на микробиота. За всеки от форматите 16S rRNA и WGS компонентът на първичен анализ произвежда вектори на характеристиките, включително относително изобилие от микробни таксони в различни рангове, както и от генни групи и метаболитни пътища съгласно номенклатурите на KEGG Orthology and Enzyme Commission (EC). Освен това някои функции се анализират по специален начин поради тяхното значение за човешкото здраве - синтез на витамини и SCFA. Тези функции се оценяват за всяка проба, като се използват подбрани пътища (Допълнителен файл 2: Фигура S2).
Работен поток на уеб услугата Knomics-Biota. Работният процес е разделен на две основни стъпки: първичен и вторичен анализ, както за ампликон, така и за метагеномни данни на WGS
Първичният анализ на 16S rRNA данни се извършва с помощта на QIIME [6], от филтриране на четене до дефиниране на OTU (оперативни таксономични единици). Съдържанието на гените се прогнозира с помощта на алгоритъм PICRUSt [7]. Данните от WGS се анализират с помощта на KneadData за качествено филтриране и HUMAnN [8] - за таксономично и функционално профилиране.
Вторичният компонент за анализ, реализиран в Python v. 3.2, включва статистически анализ на векторите на характеристиките (заедно с метаданните, ако са предвидени) и генериране на статични фигури, както и вход (във формат JSON) за интерактивни модули за визуализация. Работният процес на вторичния анализ варира в зависимост от избора на типа на отчета от потребителя (вж. Фиг. 1).
Основният отчет се генерира първоначално за всички потребителски данни. Включва проверка на качеството на „суровите” данни, оценка на относителното изобилие от таксони и функционални генни групи, както и алфа-разнообразие. Извършват се йерархично групиране, ентеротипизиране [9] и прогнозиране на метаболитния потенциал. Освен основните визуализации се предоставят интерактивни модули, включително топлинна карта, PCoA (анализ на главните координати), графика на алфа-разнообразие и мрежа за съпътстващи събития [10]. Всеки модул в рамките на Basic и други интерактивни отчети на Knomics-Biota е придружен с подробности за изпълнението (използвани алгоритъм и бази данни, стойности на контролните параметри и т.н.), така че потребителят да може да репликира резултатите независимо - както и да опише методи в нечия научна публикация.
Биоинформативните алгоритми във вторичния анализ включват метод PERMANOVA за многовариатен анализ, регресионни линейни модели и U-тест за откриване на връзки между микробни характеристики и фактори. Отклоненията се идентифицират с помощта на теста на Грубс и се отстраняват от по-нататъшния статистически анализ. Извършва се многократна корекция на теста, като се използва процедурата на Бенджамини-Хохберг.
Резултати и дискусия
Разработени са редица тръбопроводи за метагеномичен анализ. Те се различават в опциите за анализ - като предоставят само първична „сурова“ обработка на данни или разширени опции, като позволяват различни формати на входни данни (16S rRNA последователност или WGS данни). Данни за сравнение са представени в таблица 1, подчертавайки, че Knomics-Biota предоставя богат репертоар от функции, което го прави по-добър от алтернативите. Както се вижда, само Knomics-Biota и MG-RAST [11] предоставят бази данни на публикувани метагеноми за сравнителен анализ. Nephele [12], както и платформите CosmosID и One Codex предоставят подобна функционалност: „сурова“ обработка на данни, усъвършенстван статистически анализ и визуализации. Никой от тях обаче не осигурява интерактивност, позволяваща да се променят параметрите на дисплея в движение.
Knomics-Biota е направен безплатно за академична употреба. За търговска употреба е предвидено специално лицензиране. Времето на безплатния анализ зависи от броя на проектите в опашката и е вероятно да се промени по време на еволюцията на системата, но в момента анализ на типичен набор от 16S rRNA, съдържащ около 100 проби от едно изпълнение на Illumina MiSeq (като преобладаващ формат на входните данни) се обработва в рамките на няколко часа. Като цяло, приблизително 5000 от 16S rRNA проби могат да бъдат подадени наведнъж от потребител. Що се отнася до анализа на WGS, поради големия обем данни и опашката обработката може да отнеме повече време - например около няколко дни за 50–100 WGS метагенома.
Преди да започнете да качвате свои собствени данни в Knomics-Biota, е възможно да разгледате пълния набор от функции на съществуващите набори от данни. След като влезе анонимно в демо сметка, на потребителя се предоставят примерни аналитични доклади, предварително изчислени за публично достъпни метагеномни данни с метаданни от няколко мащабни проучвания, изследващи микробиома при различни състояния като рак на дебелото черво [13], възпалителни заболявания на червата [14 ] и недохранване [15], както и свързани с диетични интервенции [3]. Списъкът с външните набори от данни се актуализира редовно с новоиздадени метагеноми, свързани с човешката чревна микробиота (както и други ниши).
След като се регистрира и влезе, потребителят може да създаде проект в акаунта си и да качи „сурови“ данни - метагеномни четения във формат FASTQ, получен чрез ампликон (16S rRNA) или WGS. Когато процесът на качване приключи, потребителят може да продължи с анализа - като винаги започва с основния отчет. За разлика от другите отчети, генерацията на базовия отчет не изисква нито метаданни, нито спецификация на външен контекст. Докладът включва резултатите от проверка на качеството, профилиране на таксономичен и функционален състав на микробиотата и алфа-разнообразие. Подобни съществуващи услуги често изискват сложни стъпки за конфигуриране от потребител, предоставят само основна функционалност за анализ [6] или са силно специализирани [1]. След успешното генериране на основния отчет е възможно да се извърши разширен анализ. Основните типове отчети и тяхното съдържание са показани накратко на фиг. 1.
Една от основните функции на Knomics-Biota е възможността да анализира потребителските данни в контекста на хиляди метагеноми от публично достъпни статии, предварително изчислени с помощта на същия конвейер. Колекцията от външни набори от данни се актуализира редовно. За удобство те са подредени в колекции (контексти) според темата им. Основните теми за микробиота включват възпалителни заболявания на червата (IBD), диета, трансплантация на фекална маса (FMT), антибиотици, популации в света, болест на Паркинсон и т.н. Съответно, макар че е възможно да се сравняват собствените данни с всички метагеноми в базата данни Knomics-Biota, често е разумно анализът да се ограничи до съответния контекст - като се използва доклад за външно сравнение (без потребителски метаданни) или доклад за мета-анализ (с предоставени потребителски метаданни). Когато анализът приключи, потребителят се уведомява по имейл.
Когато информацията за членството на всяка проба в случай или контролна група бъде качена, съответният отчет за контрол на случаите става наличен - позволява да се сравняват тези набори от данни статистически и визуално - подобно на сценария за външно сравнение. Функционалността на интерактивните модули е разширена, за да позволи сравнение на състава на микробиотата между двете групи. Извършва се статистически анализ за идентифициране на съответните значими разлики. Освен основните характеристики на състава, се оценяват и сравняват специфичните за чревната микробиота характеристики и се сравняват между групите: те включват метаболитен потенциал за синтез на витамини и SCFA. Докладът за сдвоени анализи има работен процес, подобен на сценария за контрол на случая, но е модифициран, за да отчете сдвоения тип данни (например метагеномите, получени от същите субекти преди и след антибиотична терапия).
Генерира се доклад за анализ на фактори, ако са предоставени метаданни с външни/присъщи фактори. Услугата извършва многофакторен анализ, за да идентифицира значими връзки между състава на микробиотата и фактори като възраст, индекс на телесна маса (ИТМ), клинично състояние и др. Интерактивните модули са разширени, за да включват контрол върху показването на тези фактори, подпомагащи изследователския анализ. Освен това отделен тип - Доклад за времеви редове - е посветен на изследването на последователно групирани извадки, включително специфични алгоритми като анализ на стабилността на таксоните и визуализации на тези точки.
За да улесни съвместните изследвания, Knomics-Biota позволява да се коригира контрола на достъпа. По подразбиране качените данни и генерираните отчети са видими само за потребителя. Възможно е обаче да споделите някой от отчетите в глобален мащаб в режим само за преглед (използвайки постоянна връзка) или да споделите проекта частно на сътрудници, регистрирани в услугата.
Заключения
Услугата Knomics-Biota е удобен инструмент за съвместен изследователски анализ на метагеномите в контекста на публично достъпни данни. Тематичните колекции от метагеноми, фокусирани върху микробиотата при специфични заболявания и популациите в света, въздействието на диетичните и медицински интервенции са полезни за сравнителни проучвания и валидиране на данните. Освен чревната микробиота, системата е готова за обработка на метагеноми от произволна среда, позволяваща на потребителите със и без опит в биоинформатиката да получат представа за системната биология на сложни микробни съобщества.
Наличност и изисквания
Име на проекта: Knomics-Biota.
Операционна система (и): Независима от платформата.
Език за програмиране: Python.
Други изисквания: браузър, интернет връзка.
Лиценз: GNU GPL.
Всички ограничения за използване от неакадемични лица: академичната употреба е безплатна; за търговска употреба се изисква лиценз.
Съкращения
Мазна киселина с къса верига
Последователност на целия геном
Препратки
Yarygin KS, et al. Resistomap - онлайн визуализация на антибиотичен резистом на микробиота на човешките черва. Биоинформатика. 2017; 33 (14): 2205–6.
Yarygin K, Tyakht A, Larin A, Kostryukova E, Kolchenko S, Bitner V, Alexeev D. Изобилието на профилиране на специфични генни групи с помощта на предварително изчислени метагеноми на червата дава нови биологични хипотези. PLoS One. 2017; 12 (4): e0176154.
Klimenko N, et al. Отговорите на микробиома на неконтролирана краткосрочна диетична интервенция в рамките на гражданския научен проект. Хранителни вещества. 2018; 10 (5): 576.
Одинцова В, Тяхт А, Алексеев Д. Насоки за статистически анализ на данните за микробния състав, изведени от метагеномното секвениране. Curr Issues Mol Biol. 2017 г .; 24: 17–36.
Судариков К, Тяхт А, Алексеев Д. Методи за визуализация и анализ на метагеномни данни. Curr. Издава Мол. Biol. 2017; 24: 37–58.
Caporaso JG, et al. QIIME позволява анализ на данни за последователност на общността с висока производителност. Методи Nat. 2010; 7 (5): 335–6.
Langille MGI, et al. Предсказуемо функционално профилиране на микробни общности, използващи 16S rRNA маркерни секвенции. Nat Biotechnol. 2013; 8: 1–10.
Abubucker S, Segata N, Goll J, et al. Метаболитна реконструкция за метагеномни данни и нейното приложение върху човешкия микробиом. Eisen JA, изд. PLoS Computat Biol. 2012; 8 (6): e1002358.
Arumugam M, et al. Ентеротипове на микробиома на човешките черва. Природата. 2011; 473 (7346): 174–80.
Kurtz ZD, et al. Оскъдно и композиционно стабилно заключение на микробни екологични мрежи. PLoS Comput Biol. 2015; 11 (5): e1004226.
Wilke A, et al. Базата данни и порталът за метагеномика MG-RAST през 2015 г. Nucleic Acids Res. 2016; 44 (Издание с база данни): D590–4.
Weber N, et al. Nephele: облачна платформа за опростен, стандартизиран и възпроизводим анализ на данни от микробиоми. Биоинформатика. 2017; 8 (2017): 1411–3.
Zeller G, Tap J, Voigt AY и др. Потенциал на фекална микробиота за ранен стадий на откриване на колоректален рак. Mol Syst Biol. 2014; 10 (11): 766.
Halfvarson J, Brislawn CJ, Lamendella R, et al. Динамика на микробиома на човешките черва при възпалителни заболявания на червата. Природа Микробиол. 2017; 2: 17004.
Smith MI, Yatsunenko T, Manary MJ, et al. Микробиомите на червата от малавийски двойки близнаци несъответстват на квашиоркор. Science (Ню Йорк, Ню Йорк). 2013; 339 (6119): 548-54.
Работна група за НМЗ на HMP. Проектът NIH за човешки микробиом. Геном Res. 2009; 19: 2317–23.
Благодарности
Благодарим на Data Laboratory за разработването на интерактивни модули, Go4ward за разработката на уебсайта, Дмитрий Родионов и Андрей Остерман (Sanford Burnham Prebys Medical Discovery Institute) за помощ при курирането на метаболитни пътища.
Финансиране
Тази работа беше подкрепена от Фонда за развитие на Центъра за разработване и комерсиализация на нови технологии „Сколково“ [# G94/16 към Knomics LLC].
Наличност на данни и материали
Информация за автора
Принадлежности
Отдел за изследвания и развитие, Knomics LLC, Иновационен център „Сколково“, Москва, Руска федерация
Дария Ефимова, Анна Попенко, Анатолий Василев, Иля Алтухов, Никита Довидченко, Вера Одинцова, Наталия Клименко, Робърт Лошкарев, Мария Пашкова, Анна Елизарова, Виктория Ворошилова, Сергей Славски, Юрий Пеков, Екатерина Филипова, Татяна Шашкова Левгеев, Евгениев
Лаборатория за компютърни технологии, Университет ITMO, Санкт Петербург, Руска федерация
Александър Тяхт и Дмитрий Алексеев
Факултет по биологична и медицинска физика, Московски физико-технологичен институт (Държавен университет), Москва, Руска федерация
Иля Алтухов, Мария Пашкова, Анна Елизарова, Виктория Ворошилова, Сергей Славски, Татяна Шашкова и Евгений Левин
Катедра "Науки за живота", Институт за наука и технологии "Сколково", Москва, Руска федерация
Отдел по биология, Московски държавен университет "Ломоносов", Москва, Руска федерация
Институт по цитология и генетика, Новосибирски държавен университет, Новосибирск, Руска федерация
Институт за изследване на протеини, Руска академия на науките, Пущино, Москва, 142290, Русия
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Можете също да търсите този автор в PubMed Google Scholar
Вноски
AT и DA ръководиха работата. IA, AV, RL и ND са проектирали архитектурата на уеб услугата. AV, IA, DE, AT и YP ръководиха работата в екип. DE, NK, IA, AV, AP, ND, VO, RL, MP, AE, VV, SS, EF, TS и EL разработиха софтуера. NK, DE, ND, MP, AE, VV, SS и EL събират, обработват и обработват данните. AP, AT и DE подготвиха ръкописа. Всички автори са прочели и одобрили окончателния ръкопис.
- Анализ на данните за добри мазнини срещу лоши мазнини - Марти Кендъл
- Как храненето и майчината микробиота оформят новородената имунна система Nature Reviews Immunology
- Lactobacillus Rhamnosus за профилактика на диария, свързана с химиотерапия - изглед в пълен текст
- Как да укрепим храносмилателната система чрез Аюрведа - Аюрведа Банско
- Първи стъпки в LCHF; Част 1 Ден на почистване Науката за човешкия потенциал